技术专题
利用iTRAQ定量蛋白质组学筛选与DAI相关的生物标志物
文章来源:genecreate
作者:genecreate
发布时间:2018-09-05 16:53
Itraq定量蛋白质组学技术和生物信息学分析鉴定大鼠弥漫性轴索损伤的血浆标志物
题目:Identification of plasma biomarkers for diffuse axonal injury in rats by iTRAQ-coupled LC–MS/MS and bioinformatics analysis
期刊:Brain Research Bulletin
影响因子:3.440
合作技术:iTRAQ
研究背景
DAI(弥漫性轴索损伤)是一种严重且复杂的脑损伤,目前临床上没有相关可靠的生物标志物帮助其进行早期DAI诊断。因此,作者建立了DAI大鼠模型,并利用iTRAQ定量蛋白质组学技术筛选与DAI相关的血浆生物标志物,该研究既有助于DAI临床诊断的发展,也对DAI潜在分子机制有了进一步了解。
研究内容结果
1. 大鼠脑组织病理学检查
作者采用HE染色法和Bielschowsky神经纤维银染法对构建的DAI大鼠模型(1h、6h、1d、3d、7d)进行脑组织病理学检查 ,结果显示对照组脑组织样本未发生任何类型的病理改变,模型组的胼胝体中则观察到不同程度的轴突肿胀、紊乱、断开及轴缩球(图1)。这些结果证实,研究中使用的大鼠样本适合于进行iTRAQ的定量蛋白质组学分析以鉴定大鼠中的DAI生物标志物。
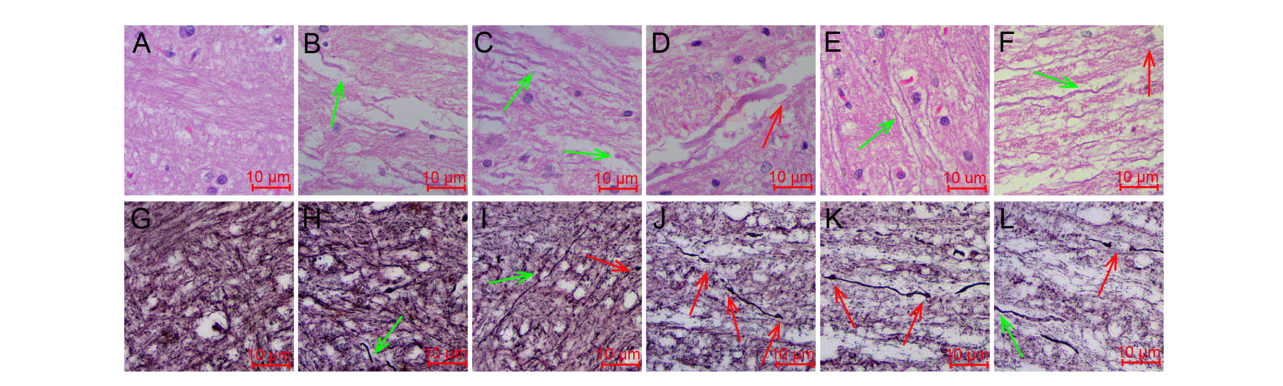
图1 大脑组织样本HE染色(A-F)和Bielschowsky神经纤维银染色(G-L)结果
2. DAI大鼠模型蛋白质组学结果
在对血浆样品进行iTRAQ蛋白质组学分析中,共鉴定到374个蛋白质,其中共有58个显著差异蛋白(FC > 1.5,p-values<0.05),和对照组相比,不同时期的模型组中差异蛋白数目如图2。针对这些差异蛋白,作者进行了GO功能分析和KEGG Pathway分析(图3)。根据58个差异蛋白的层次聚类分析结果,在3d和7d中的蛋白质丰度发生显著改变(图4A)。这可能是因为DAI的发病机制复杂,涉及多阶段的生物学过程,轴突损伤1d后可能与不同的分子机制有关。为了研究不同差异蛋白的相互作用,作者又进行了蛋白互作网络分析,结果显示GAPDH和Hpx等几种蛋白质位于蛋白质功能相互作用网络的中心位置(图4B)。
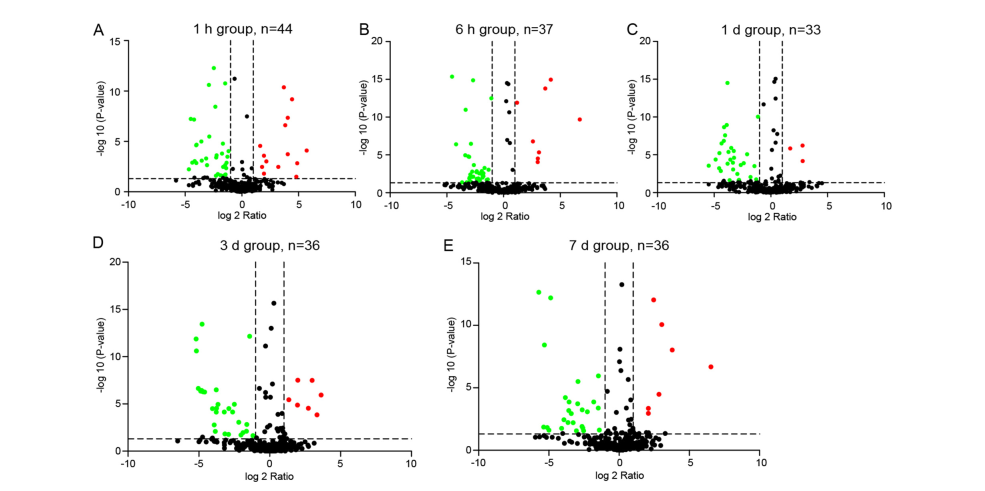
图2 不同DAI时期蛋白表达火山图
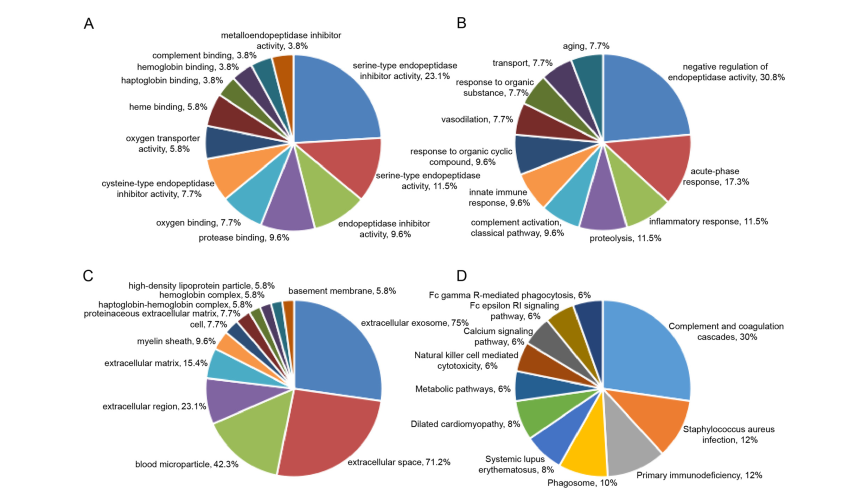
图3 差异蛋白GO功能分析和KEGG Pathway 分析
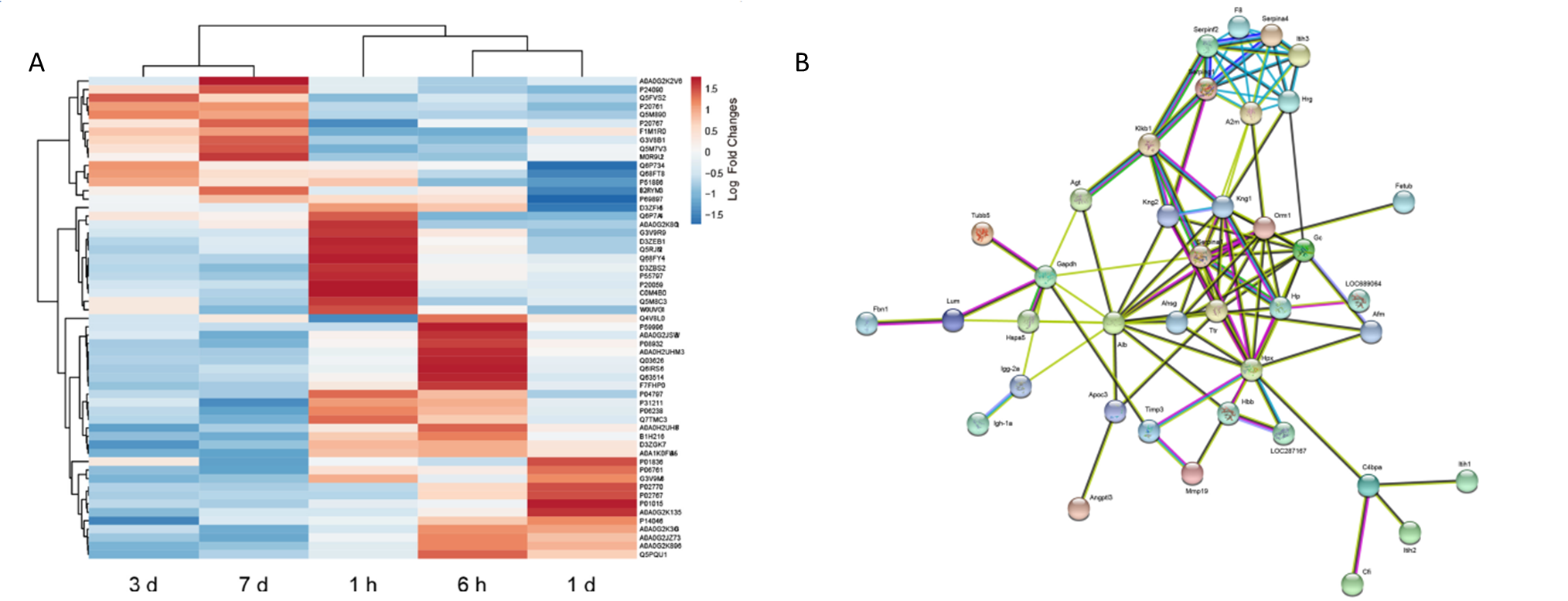
图4 差异蛋白层次聚类分析和蛋白互作网络分析
3. 候选标志物验证
基于蛋白质组学结果,结合蛋白在DAI期间轴突损伤过程中的表达量发生极大变化和潜在的生物学功能,作者选择GAPDH和Hpx作为候选标志物。和对照组相比,Hpx的表达量在损伤后6h、1d、3d、7d中都显著下调;GAPDH表达量在损伤后1h、6h显著上调,随后又在1d、3d、7d中显著下降。WB结果与蛋白质组学结果一致(图5)。
Hpx是一种丰富的血浆糖蛋白,对血红素有极高的亲和力,已有研究证明敲除Hpx会加重无基质血红蛋白诱导的脑损伤,动物实验和人神经母细胞瘤细胞培养实验均显示Hpx提供针对血红素活性氧(ROS)毒性的神经保护作用。脑损伤后Hpx表达降低可能加重DAI患者的脑损伤,最终导致死亡。在本研究中,DAI模型的Hpx表达水平在6h、1d、3d和7d组中显著低于对照组,而在1h组中没有显着变化,这意味着Hpx可能参与了非断离性轴索损伤的生物学过程。这些发现还表明,在DAI中促进Hpx的表达可能有助于减少轴突断裂和提高临床疗效。
GAPDH是参与糖酵解过程的重要酶,已有研究报道TBI(创伤性脑损伤)后GAPDH显着降低,同时也会导致能量需求增加。脑损伤后GAPDH显着下调可导致能源供应的早期减少,最终导致能源供需之间的不平衡。但是下调的GAPDH是否参与非断离性轴索损伤中的分子和细胞级联仍有待确定。这些研究结果表明,适当改善能量供应可能有助于减少轴索损伤并获得更好的DAI患者预后效果。
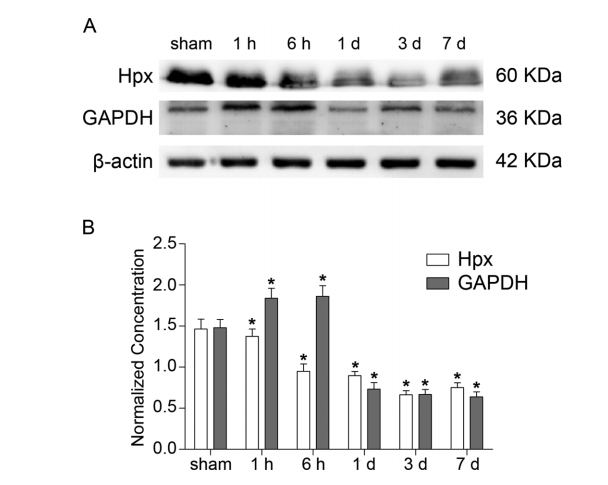
图5 western blot 验证候选标志物
研究结论
在本研究中,作者构建了DAI大鼠模型,并应用iTRAQ定量蛋白质组学方法筛选与DAI相关的差异表达的血浆蛋白。基于蛋白质组学结果和蛋白的生物学功能,作者筛选得到了两个蛋白(Hpx和GAPDH)作为潜在的诊断生物标志物,它们可能在DAI的轴突损伤的生物过程中发挥重要作用。作者的研究结果不仅为DAI的分子机制提供了新的见解,而且还促进了DAI临床诊断生物标志物组的开发。
解析文献
Peng Zhanga, Shisheng Zhu, et al. Identification of plasma biomarkers for diffuse axonal injury in rats by iTRAQ-coupled LC–MS/MS and bioinformatics analysis. Brain Research Bulletin, 2018 , 140.
参考文献
1. Opii, W.O., Nukala, V.N., et al. Proteomic identification of oxidized mitochondrial proteins following experimental traumatic brain injury. J. Neurotrauma.2007, 24 (5), 772–789.
2. Ren, J., Zhao, G., et al. Identification of plasma biomarkers for distinguishing bipolar depressionfrom major depressive disorder by iTRAQ-coupled LC–MS/MS and bioinformatics analysis.Psychoneuroendocrinology .2017, 86, 17–24.
3. Cheow, E.S.H., Cheng, W.C., et al. Myocardial injury is distinguished from stable angina by a set of candidate plasma biomarkers identified using iTRAQ/MRM-based approach. J. Proteome Res. 2018, 17 (1), 499–515.
4. Zhang, P., Zhang, L., et al. Quantitative proteomic analysis to identify differentially expressed proteins in the myocardium of epilepsy using iTRAQ coupled with nano-LC–MS/MS. J. Proteome Res. 2018, 17 (1), 305–314.