技术专题
教你使用谱图标峰小工具—pLabel
文章来源:genecreate
作者:genecreate
发布时间:2023-02-22 09:38
在LC-MS/MS蛋白质鉴定实验中, 由于化学污染、电气噪声、低质量的准确性, 以及意料之外的翻译后修饰(PTM) 和MS / MS碎片模式, 使用搜索引擎进行大批量解析谱图过程,可能导致肽段鉴定结果存在大量假阳性。另外,搜索引擎算法上存在的缺陷也会造成肽段-谱图匹配(Peptide Spectrum Maches,PSMs)的错误。因此,人工验证PSMs质量成为必要,尤其对于特定的研究目的(如多肽鉴定,目标蛋白识别等)或特定蛋白,这时候就需要我们用自己的火眼金睛来判断谱图与给定肽段序列的匹配情况了。除此之外,有些杂志也要求提供特定肽段的PSM峰图。
今天给大家推荐一款关于谱图标峰实用的小工具——pLabel,并手把手教会大家如何使用。
1. pLabel介绍
pLabel™ (当前版本,v2.4.05)是一款专门用于蛋白质组学研究的质谱峰标注软件(mass spectral peak labeling software)。主要提供如下功能:
1)支持批处理,质谱数据和搜索引擎的搜索结果可同时直接导入;
2) 支持多种质谱数据格式(mgf,dta,ms2),搜索引擎检索结果(pFind,Mascot,SEQUEST);
3)支持ECD/ETD碎裂模式的谱图标峰;
4)根据用户定制的片段离子类型,在已知的PSM中突出显示不同颜色的匹配峰(如b离子峰为绿色、y离子峰为红色)
5)可同时实现多个氨基酸位点,多种翻译后修饰的标峰;
6)支持中性缺失标峰功能;
7)支持相关统计功能;
8)输出图片支持多种格式(png,bmp,tiff,jpg),可以单张导出或批量导出。
2、资源获取及安装
从下面链接下载Find软件:http://pfind.org/software/pLabel/。解压后找到plabel文件夹,找到plabel.exe,然后双击,开始安装。注意,pLabel目前只支持Windows环境系统。
3、软件使用
1)质谱原始文件导入(step1)
本文以MGF格式文件为例(gel_ID_002_MGFPeaklist.mgf):
打开软件,导航栏单击FILE按钮;
Load MS/MS File→选择MGF格式类型→导入MGF文件。
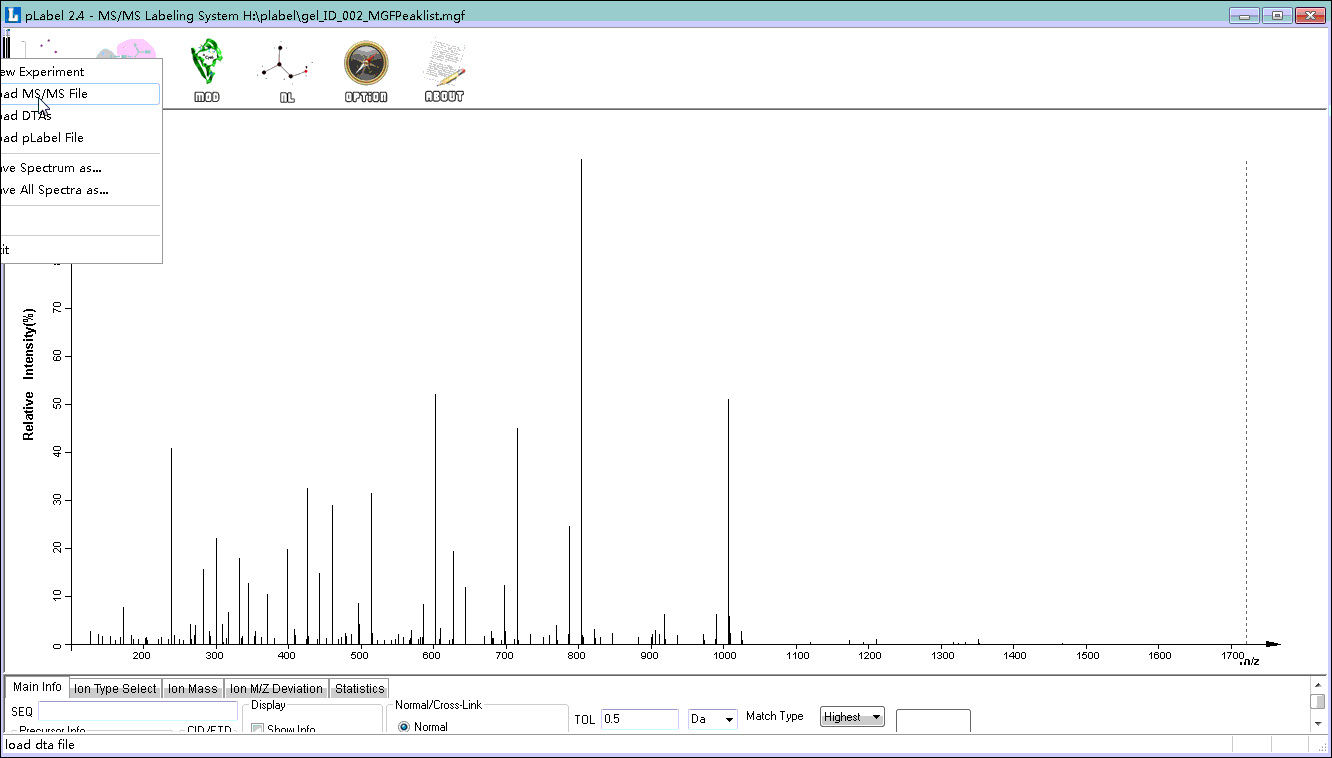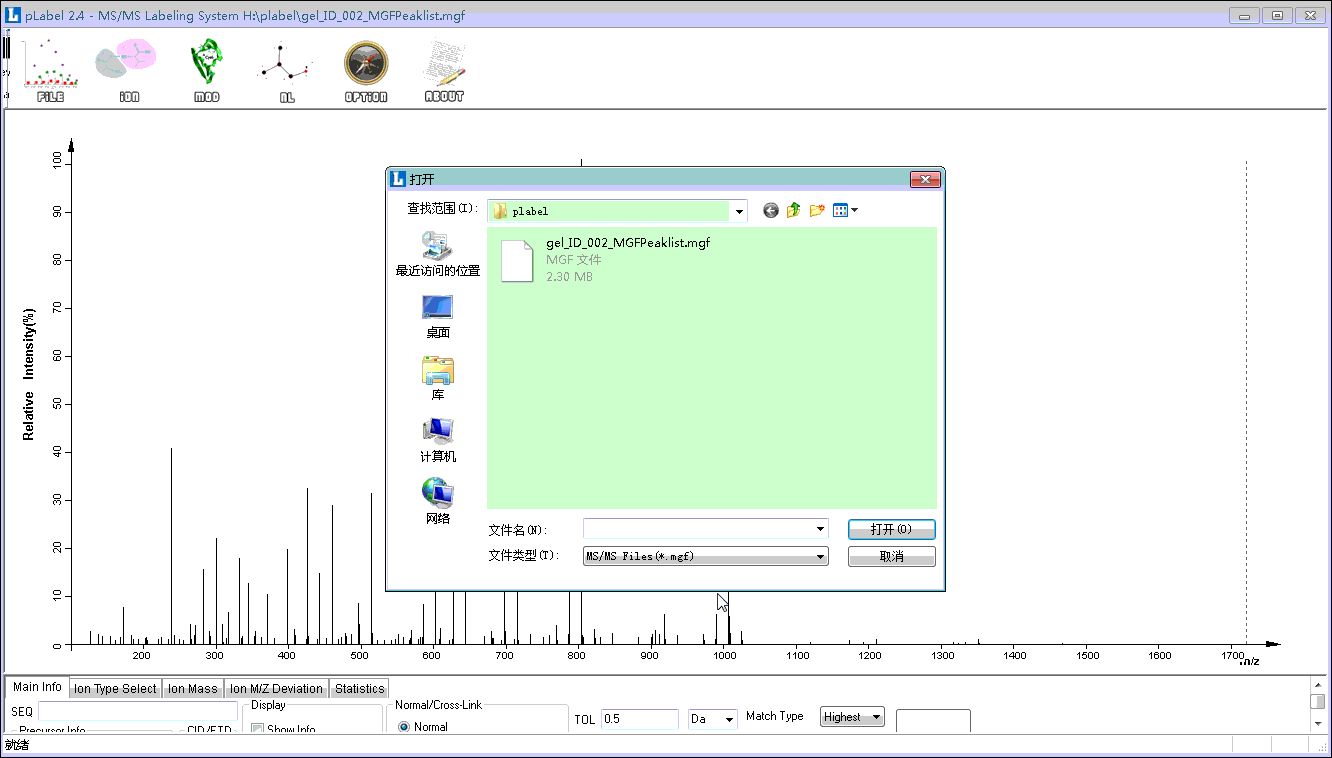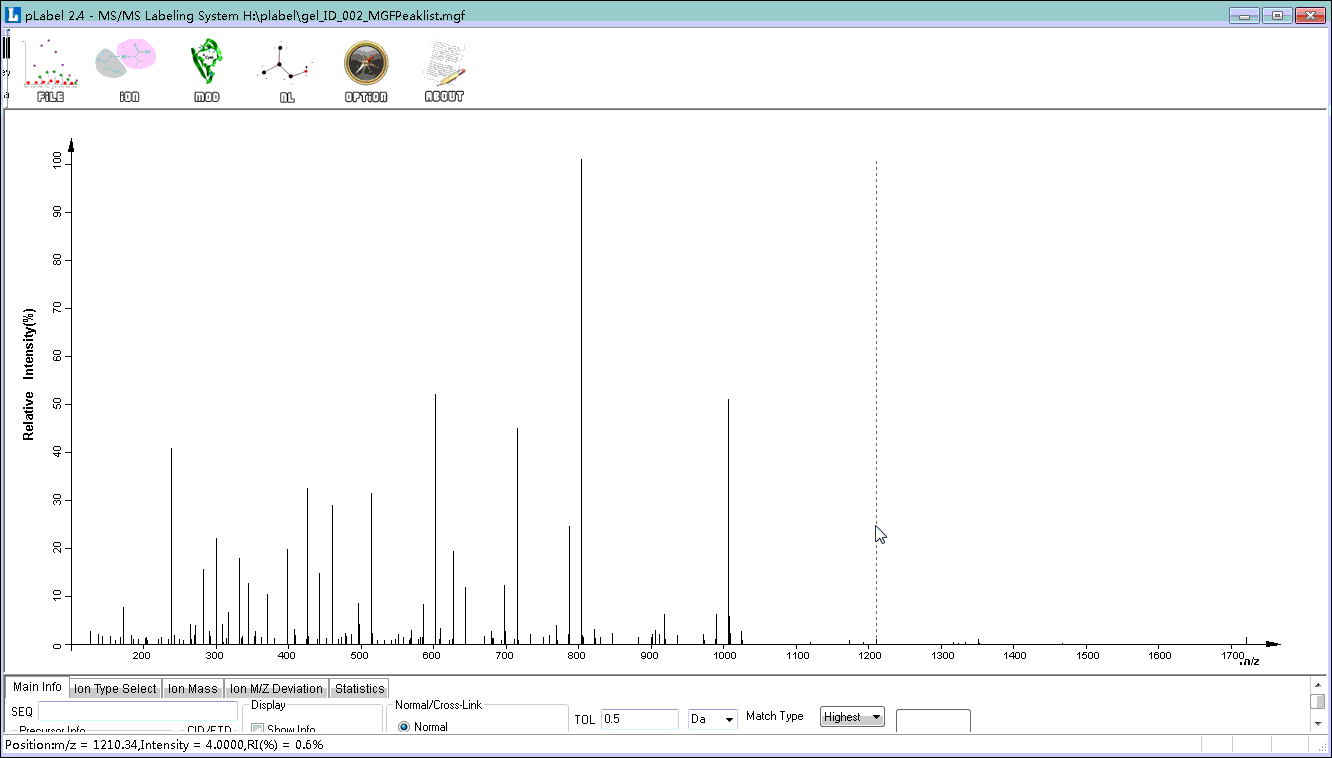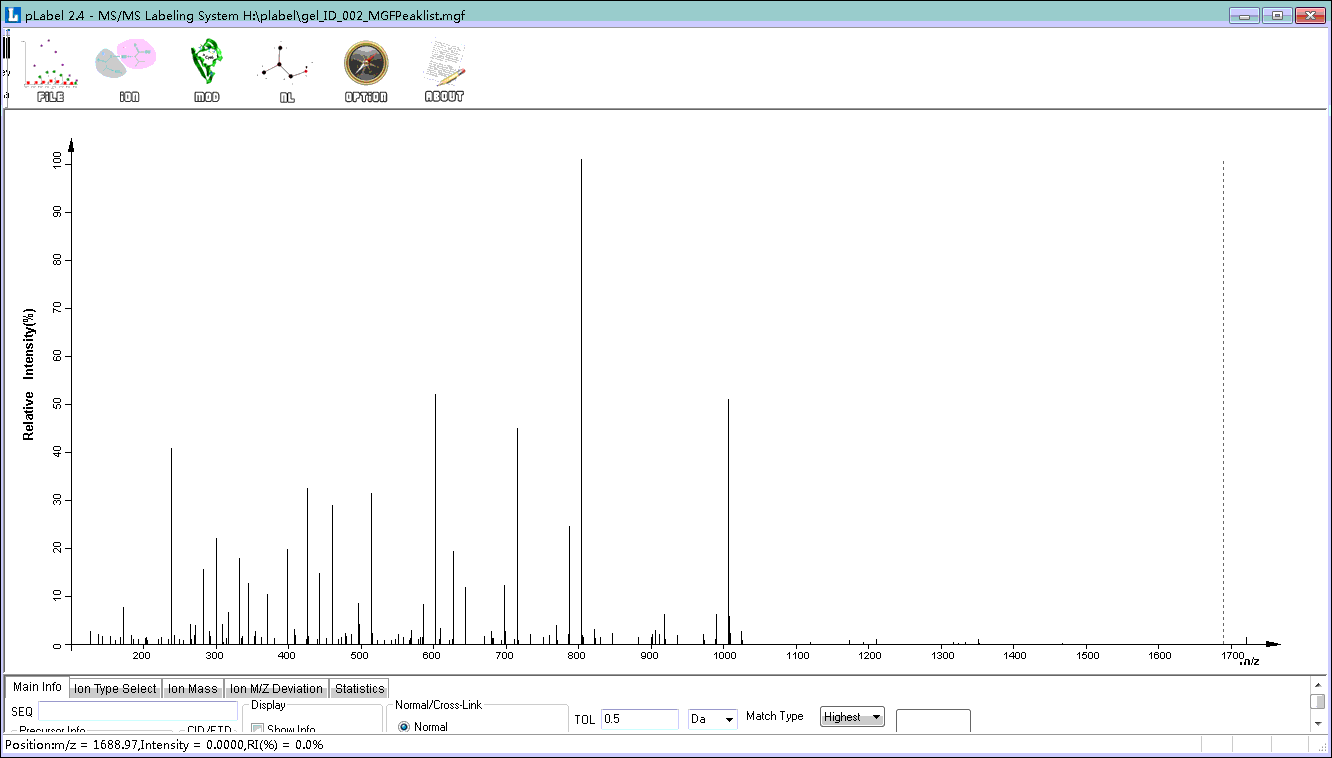
Step 1. MGF文件导入
这一步相当于将对应mgf文件中的所有二级谱图导入了plabel中。
2)查看所有谱图和指定谱图
点击界面右侧的All按钮,会弹出mgf文件中的所有二级谱图的title
在弹出的界面中选择一个,或者在左下角输入所关注的二级谱的title,然后点击确定,此时plabel界面就会显示对应的二级谱图。例如,本文想观察谱图编号为1.1.1.1109.3的二级谱。关于怎么知道所关心的谱图编号(或title),请继续往下看。
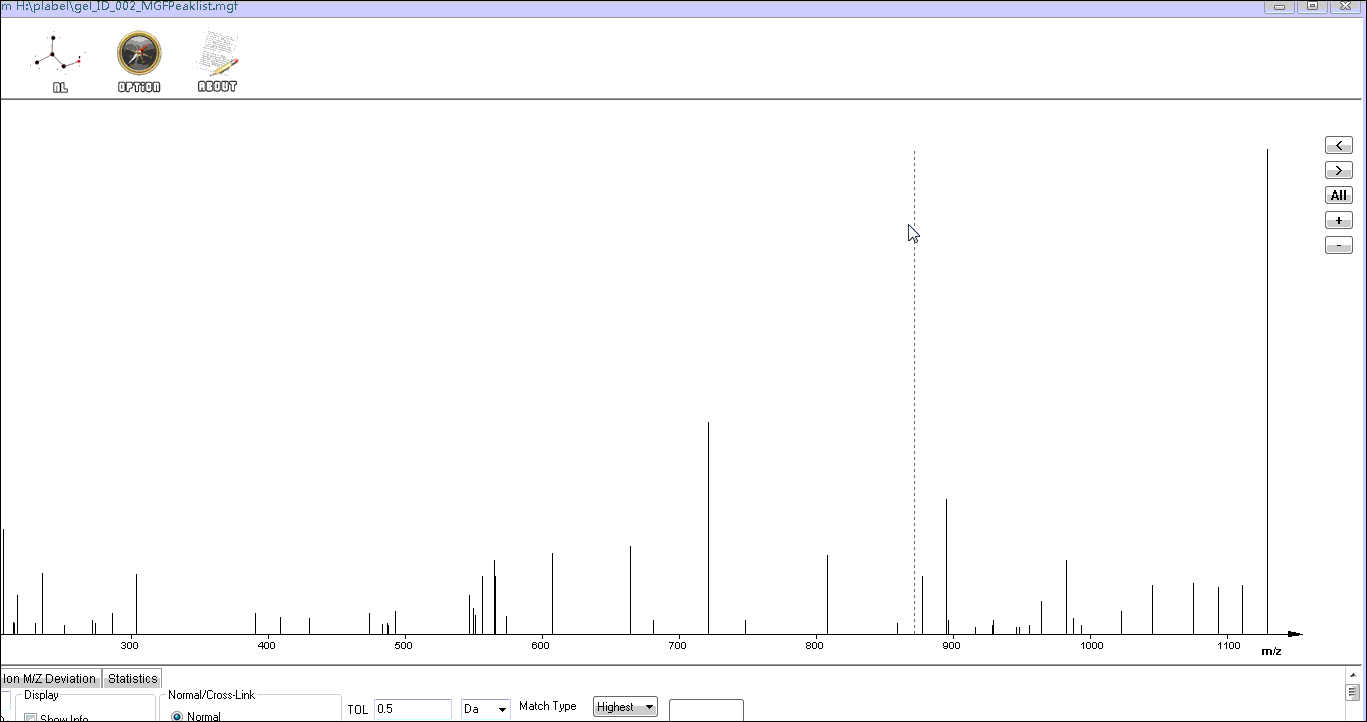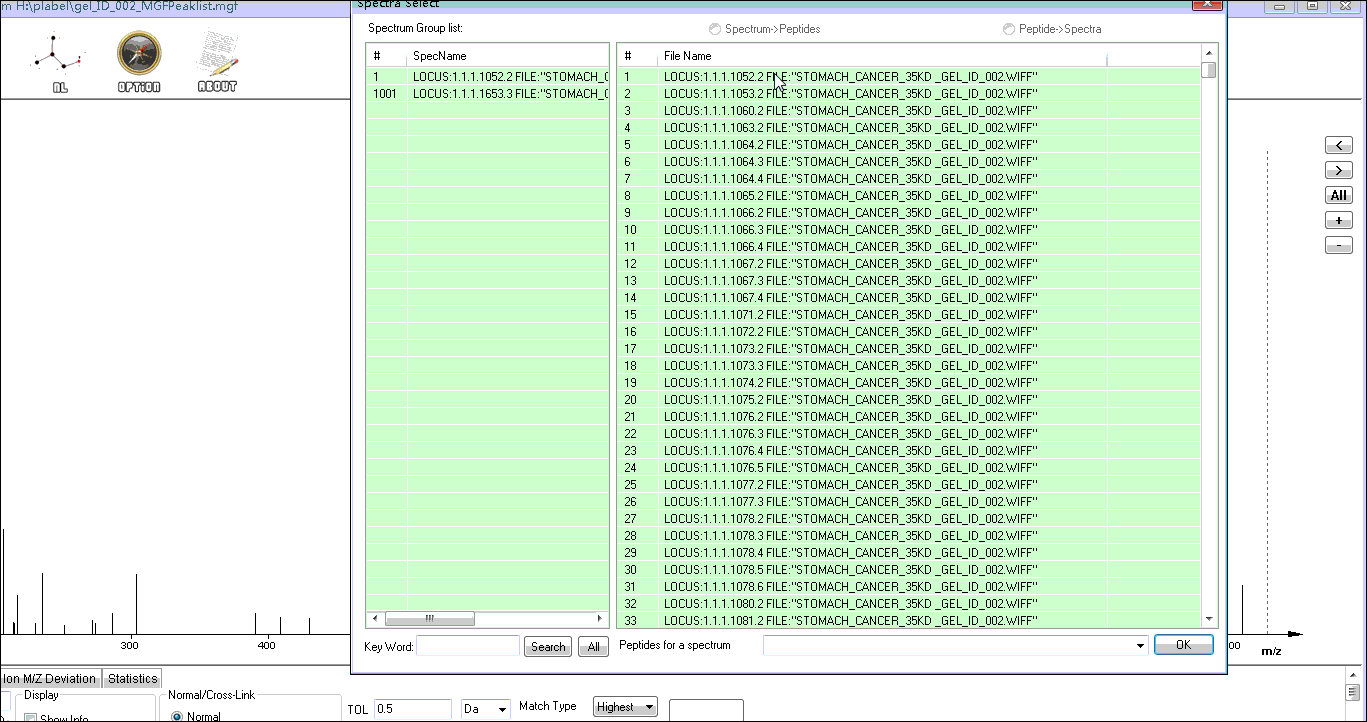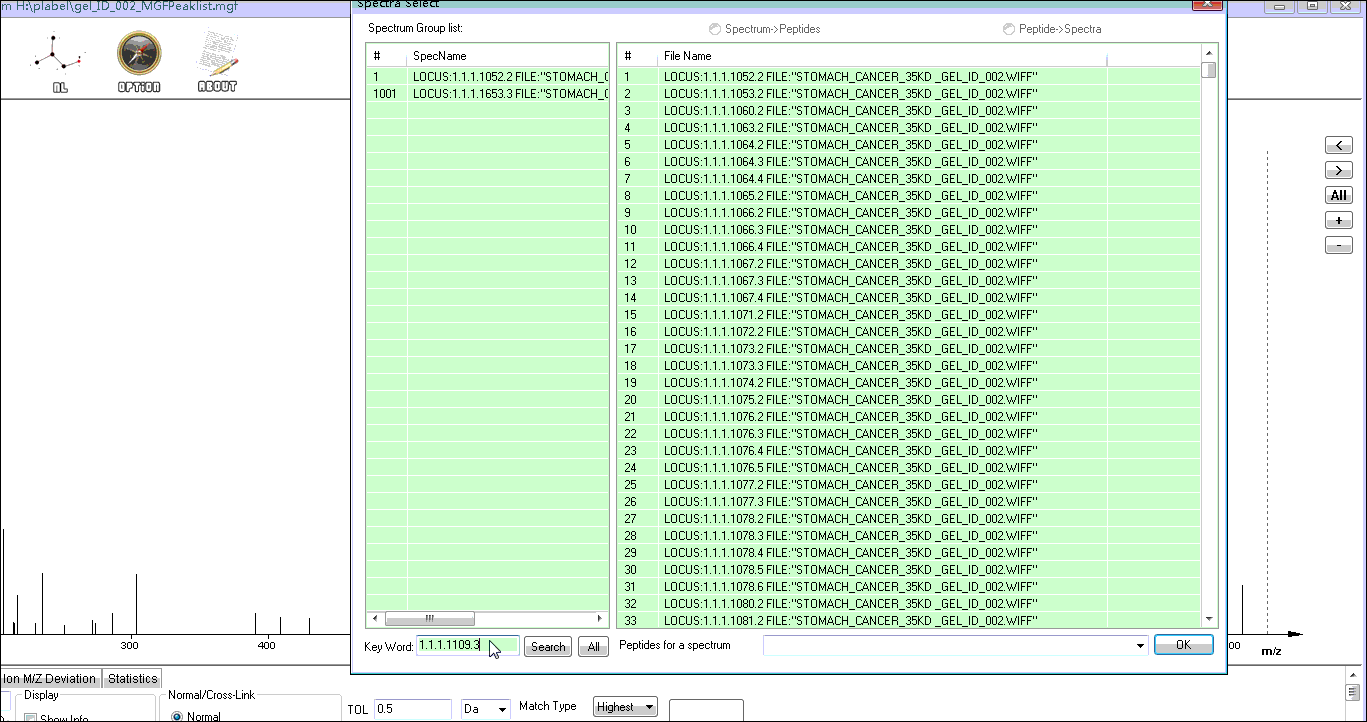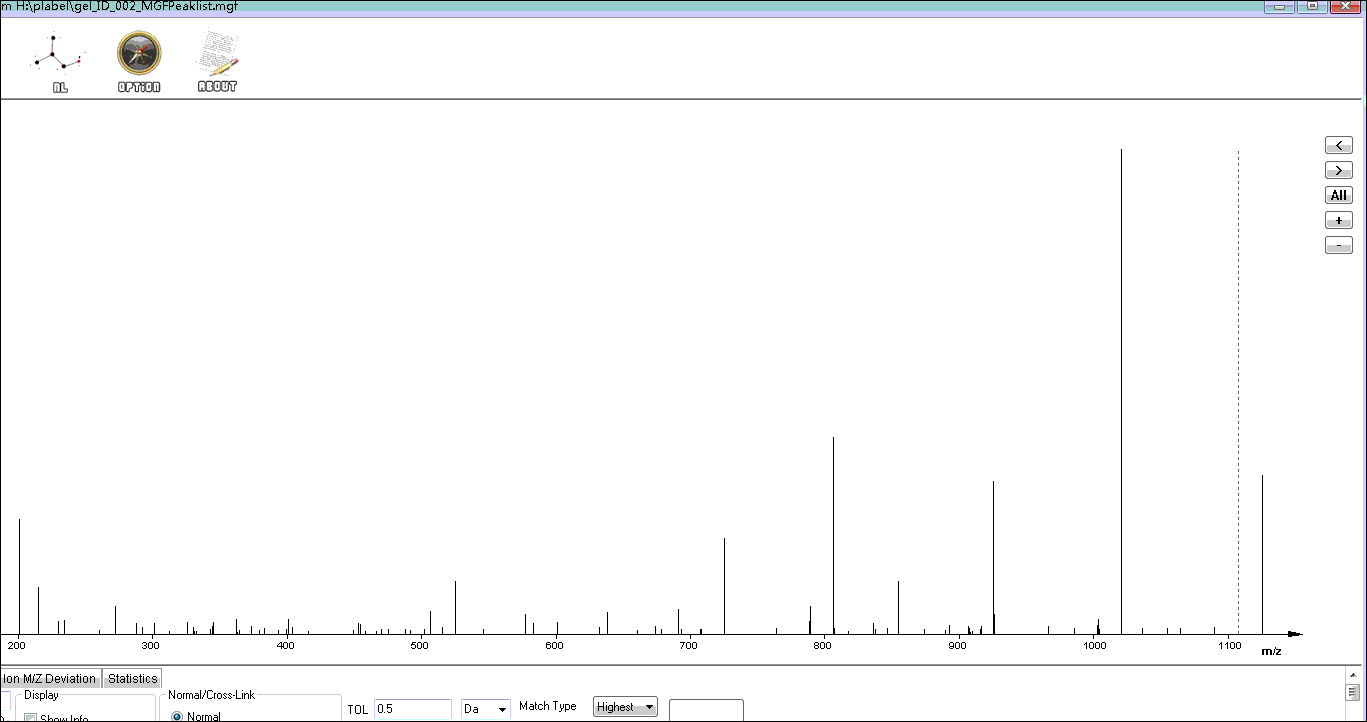
Step2. 选择特定谱图
3) 选择特定谱图-肽段,并对其进行谱峰标注
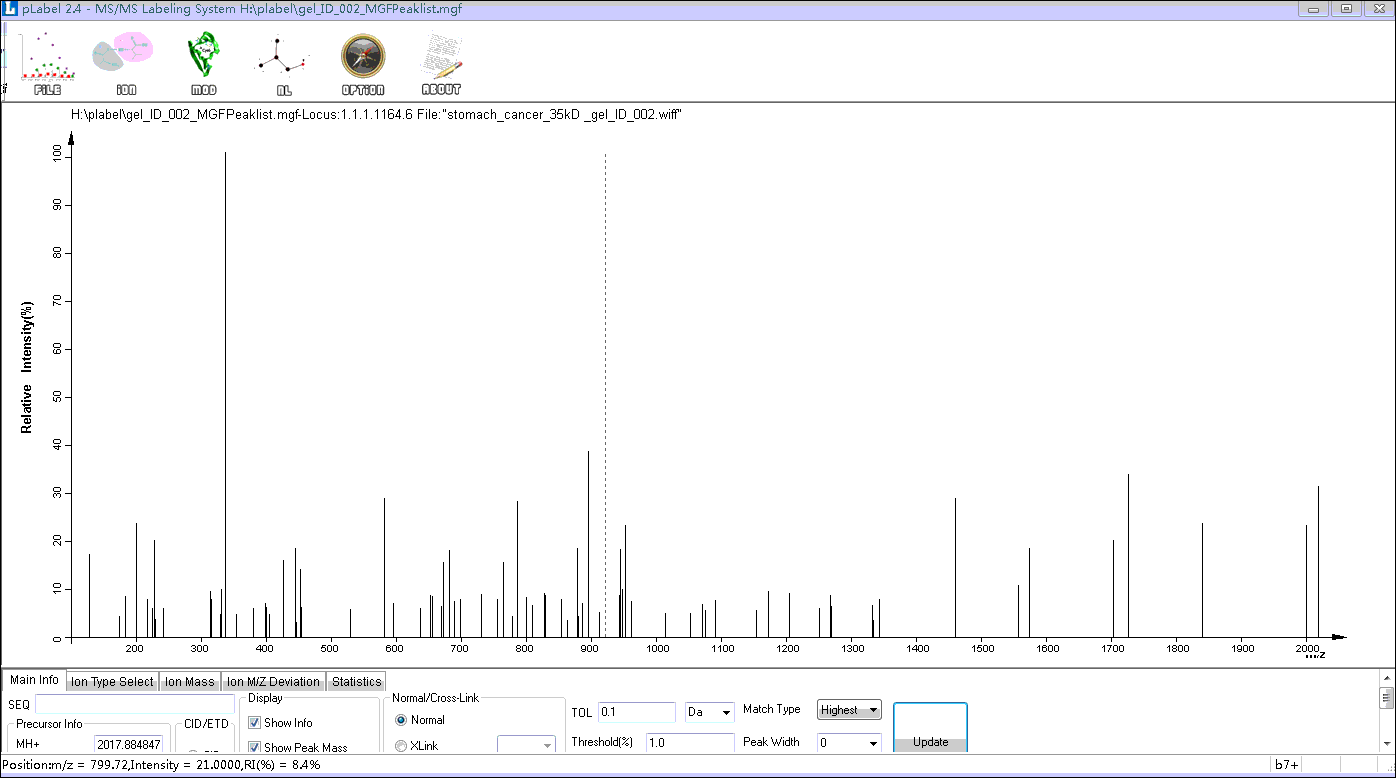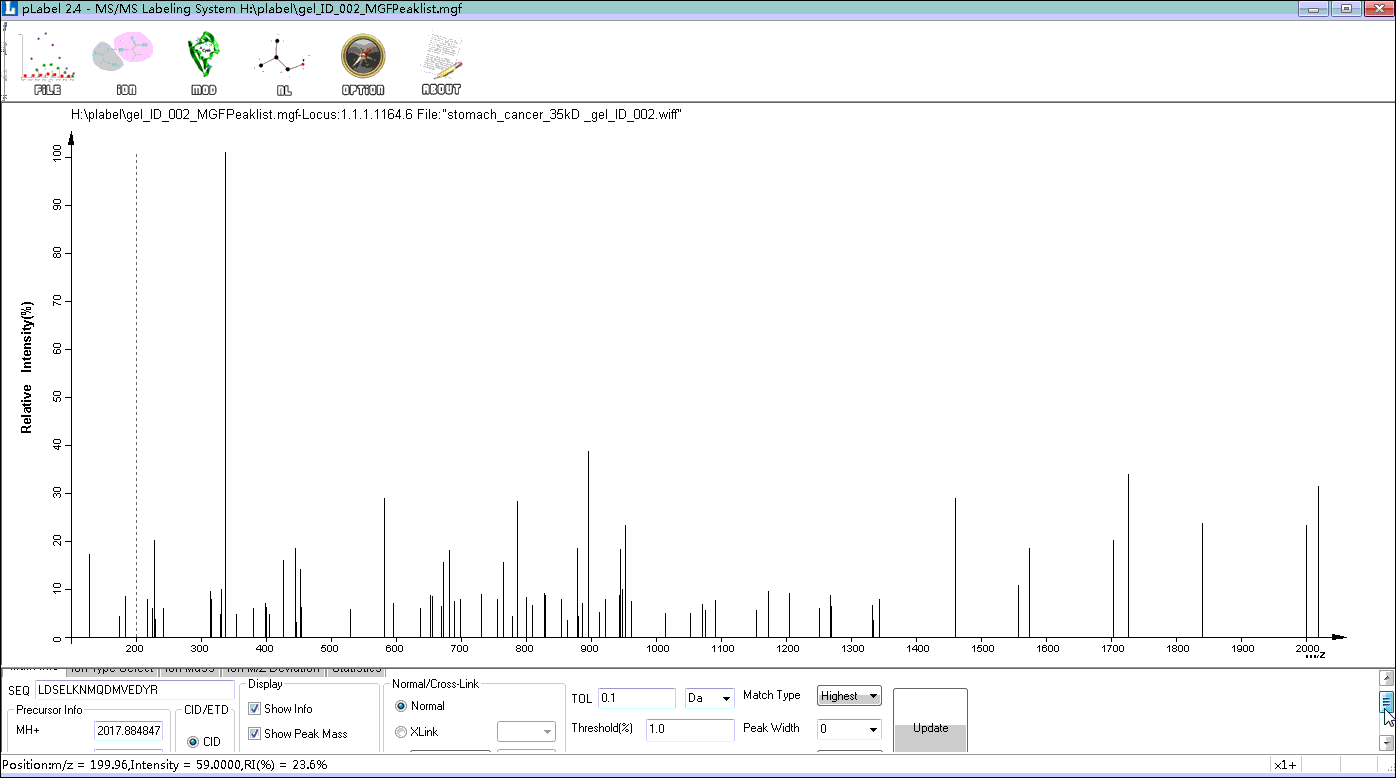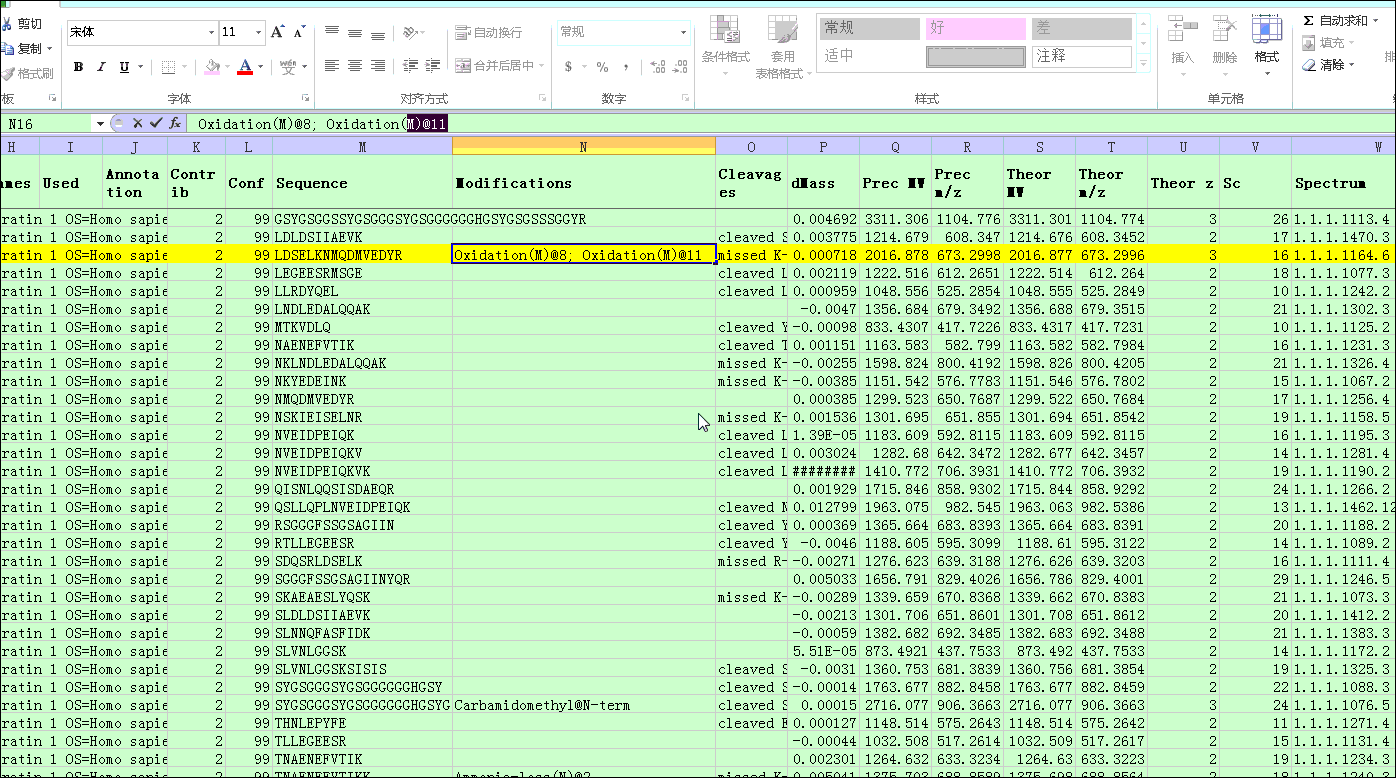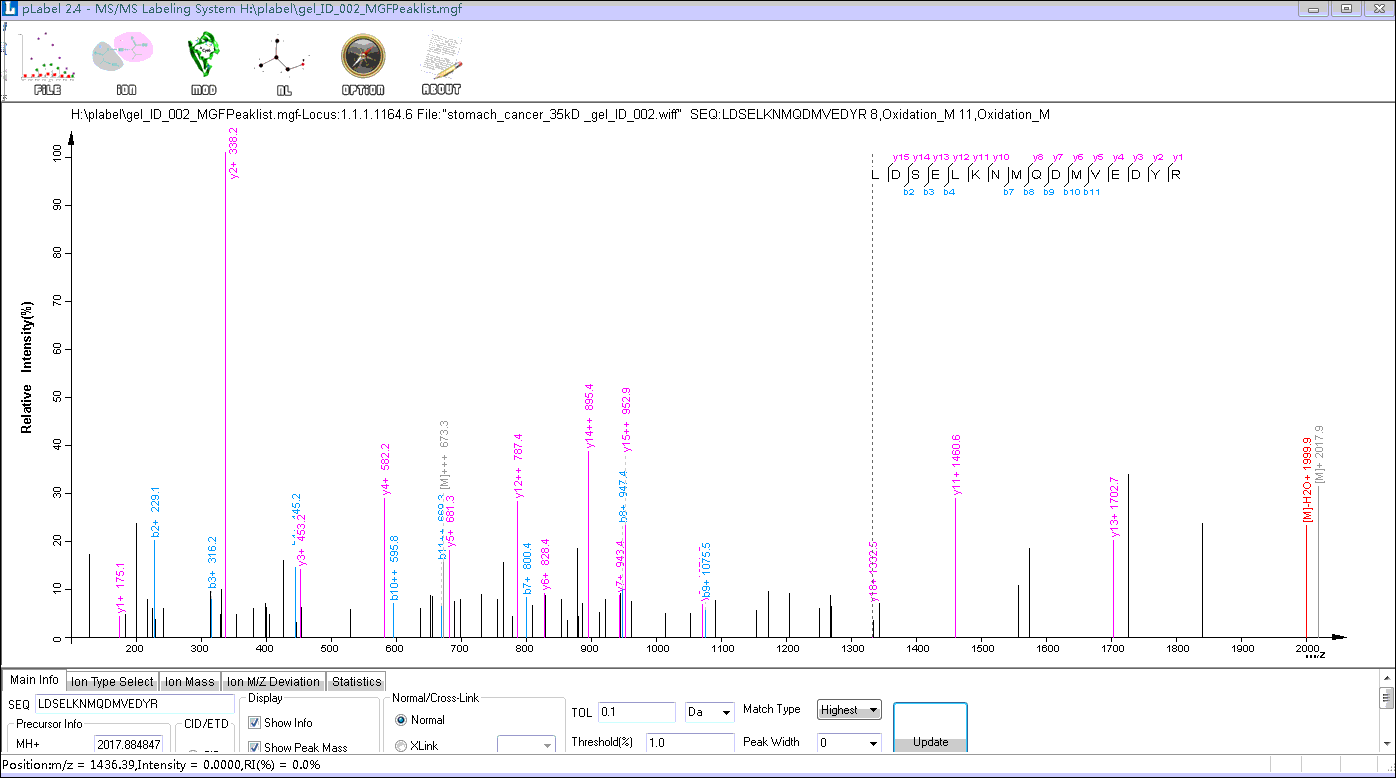
本文以Proteinpilot结果为例(如图1)。谱图编号为1.1.1.1164.6,肽段序列为LDSELKNMQDMVEDYR,可变修饰为8号氨基酸的Oxidation(M)和11号氨基酸的Oxidation(M)。
按照2)步骤选择谱图1.1.1.1164.6。
将肽段LDSELKNMQDMVEDYR复制粘贴到界面左下角的SEQ栏。
设置参数。勾选Show Info,勾选Show Peak Mass,勾选Show Neutral Loss,勾选Show AA sequence,TOL:0.1,Threshold:1.0。其它参数默认。
在对应位点添加可变修饰(单击左上角的mod选项)。
点击Update。此时界面出现了谱图的标峰图
右击图片界面空白处(图2),保存图片。
至此,对特定谱图-肽段的的标峰完成了。

图. ProteinPilot软件输出结果示例
Step3. 谱图肽段标峰
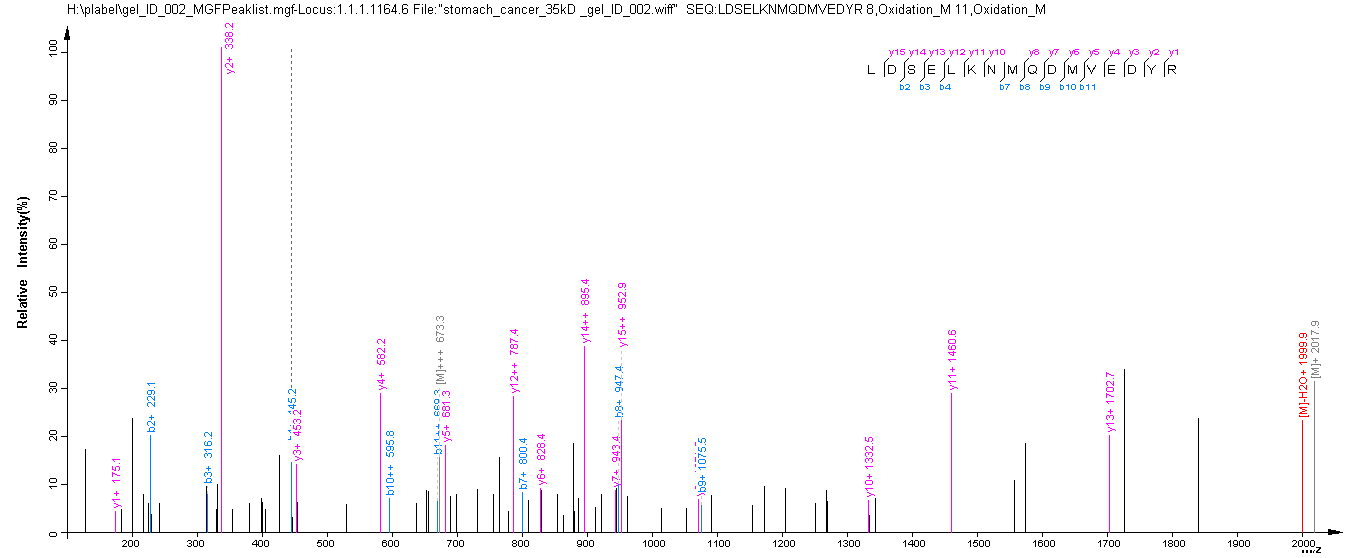
标注的谱峰图
4)高级设置
自定义需要标注的碎片离子类型
默认下,plabel只标注-b和-y系列离子。我们可以根据质谱仪的型号与采集模式标注其他类型碎片离子。方法如下(step4):
点击下方导航栏中的Ion Type Select按钮;
选择需要添加的离子类型。对大多数质谱仪将肽段母离子碎裂成b、y系列离子(一价或二价),并且有中性丢失的情况发生。因此,我们添加b、y的中性丢失;
点击下方导航栏的Main info返回设置主界面。点击Updat,会发现在标峰图中多了相应的谱图标注信息。
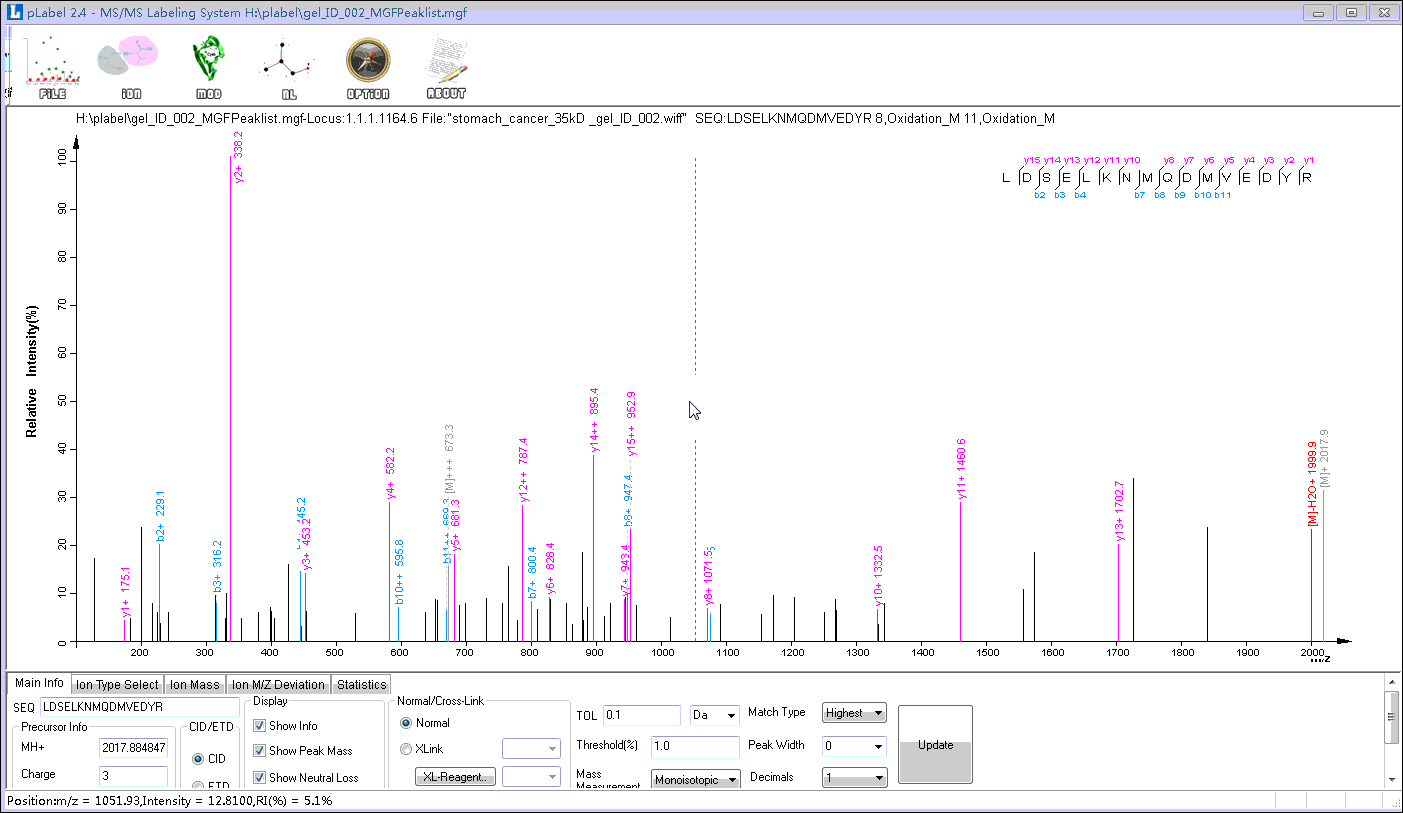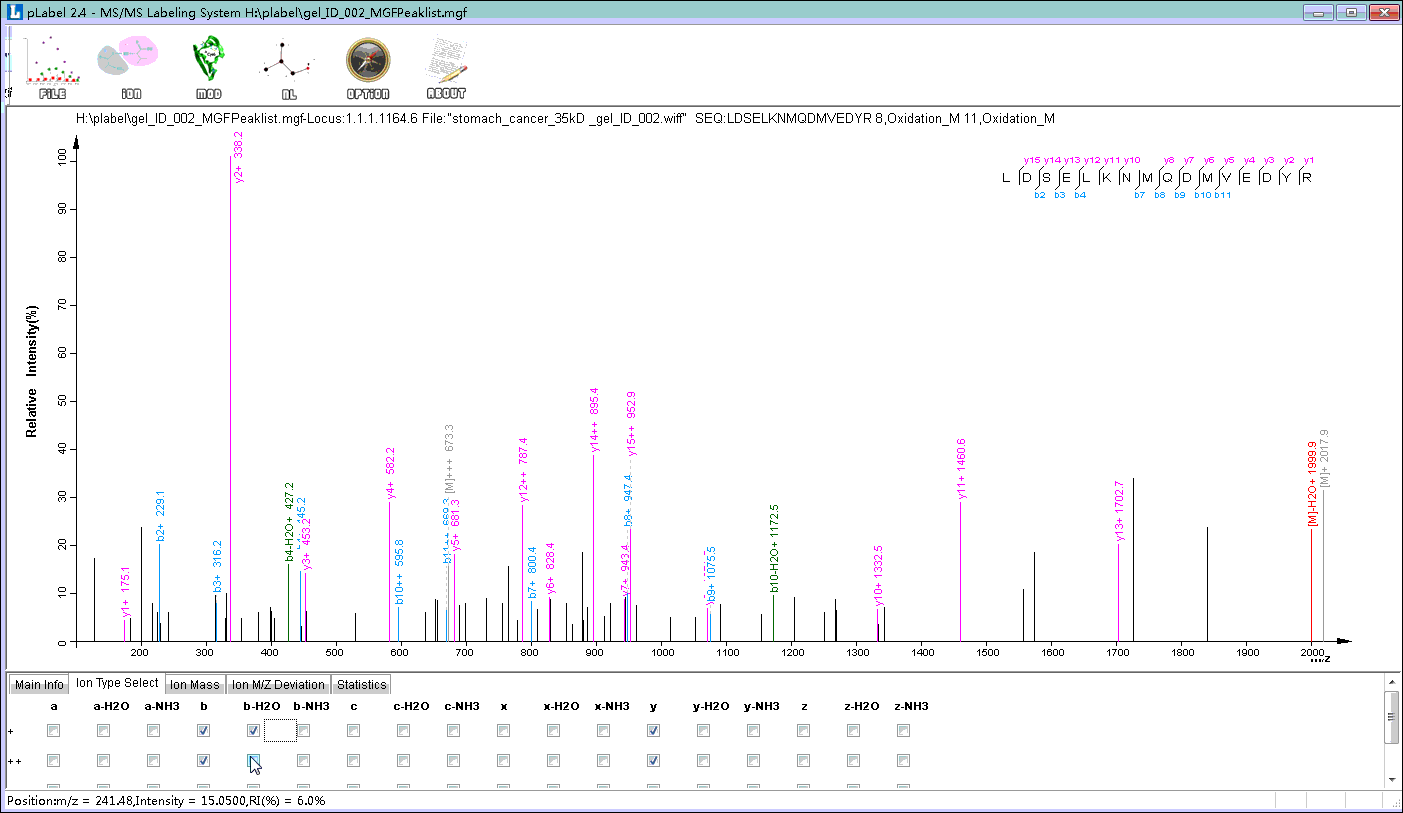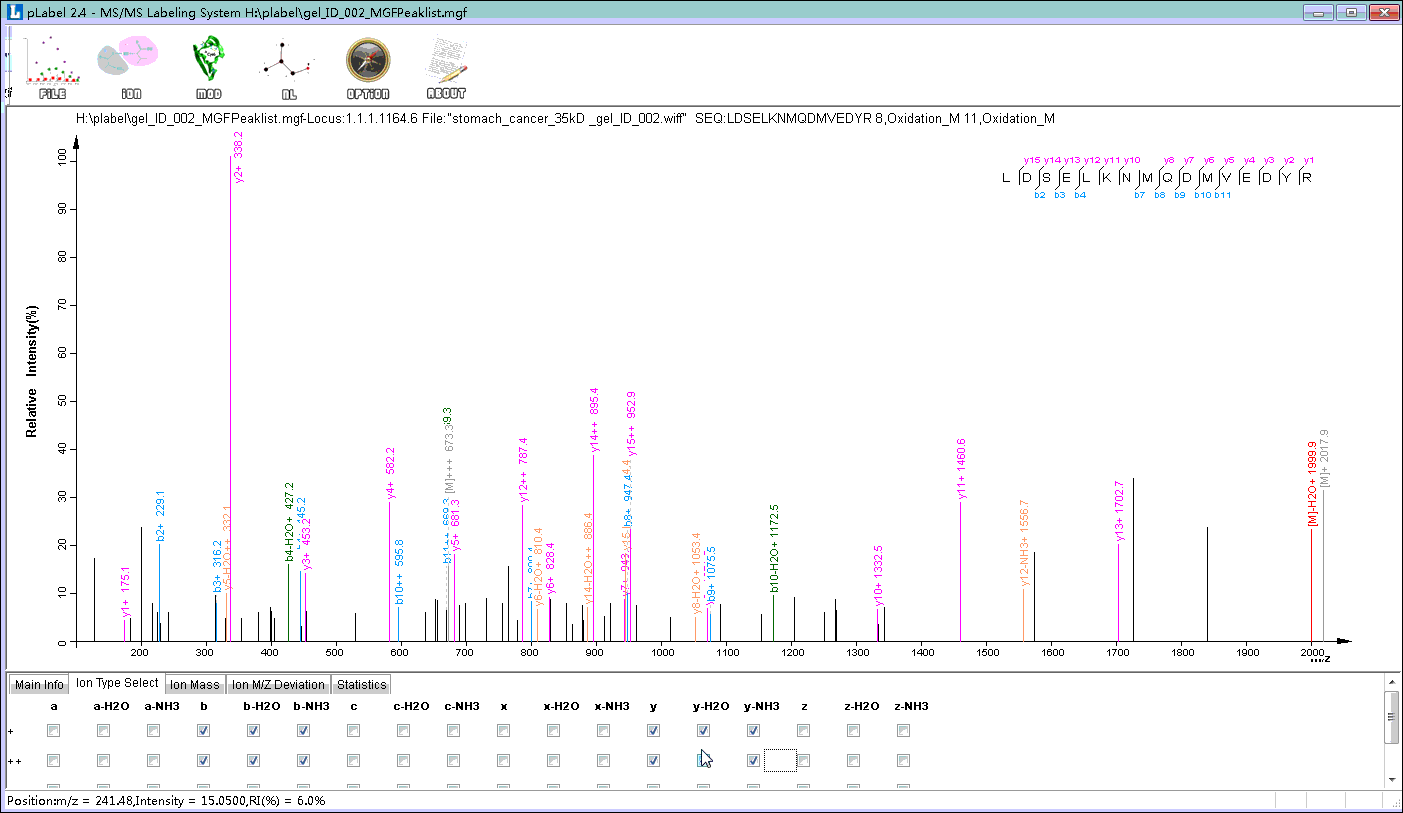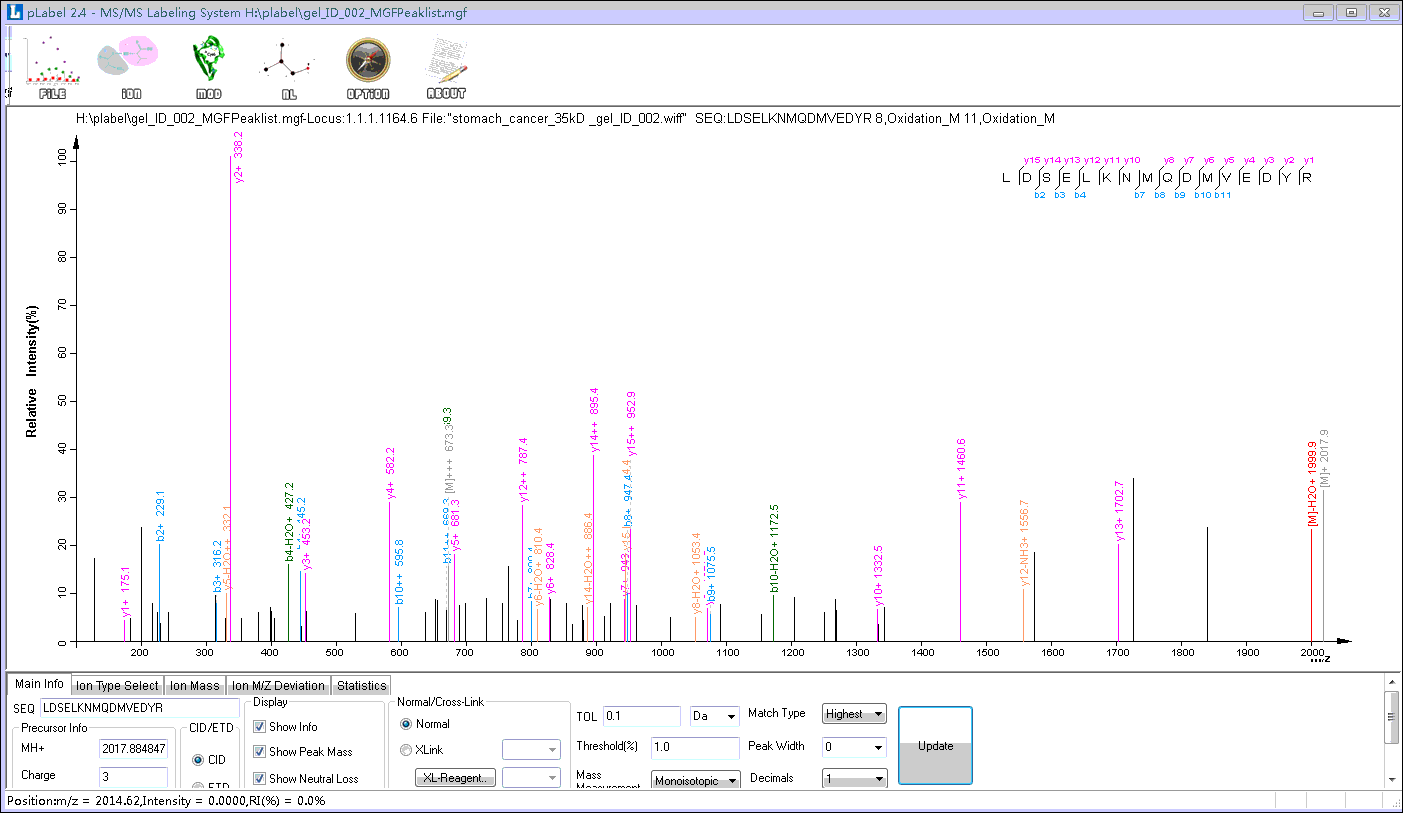
Step4. 选择其他类型的碎片离子
自定义修饰类型
有时候,默认的plabel中的修饰类型不包含想要添加的类型。这时候就需要我们自己动手添加了。以谱图1.1.1.1114.2和对应的肽段AQYEDIAQK为例,其修饰类型为:Methyl(D)@5; Formyl(K)@9。发现默认的plabel中没有这两个修饰。在添加新修饰类型之前我们需要确定以下信息:修饰基团的分子量(单一同位素分子量和平均同位素分子量)、修饰位点和中性缺失。这些信息可以直接在对应的搜索引擎中寻找,也可以到unimod官网上查看(http://www.unimod.org/modifications_list.php),如下,
Unimod查找:
进入网页,以guest身份登录;
将修饰名称(Methyl)输入到搜索栏,点击Search按钮;
在出现的结果中找到Methyl,ID为34,点击View进入该修饰界面;
可以看到,Methyl修饰的化学式为H(2)C,单一同位素(Monoisotopic)分子量为14.015650,平均同位素(Average)分子量为14.0266,无中性丢失。可发生Methyl修饰的氨基酸包括C,H,K,N,N-term,Q,R,I,L等。
将Methyl修饰添加到plabel中(step5):
按照前述步骤选择谱图1.1.1.1114.2,输入肽段AQYEDIAQK
点击plabel上方主导航栏中的ion按钮;
选择Mass Config→Modification→Add,此时会弹出添加修饰类型的小窗口;
添加自定义修饰。Name为自定义名字,最好能够顾名思义,以方便以后使用。本示例输入Methyl(D);Site为发生修饰的位点,输入D;Modification Type为修饰类型,为NORMAL;Mass(Mono):14.015650,Mass(Avrg):14.0266。点击OK保存;
同样的方法添加Formyl(K)修饰;
按前述步骤将Methyl(D)@5; Formyl(K)@9添加到序列中。
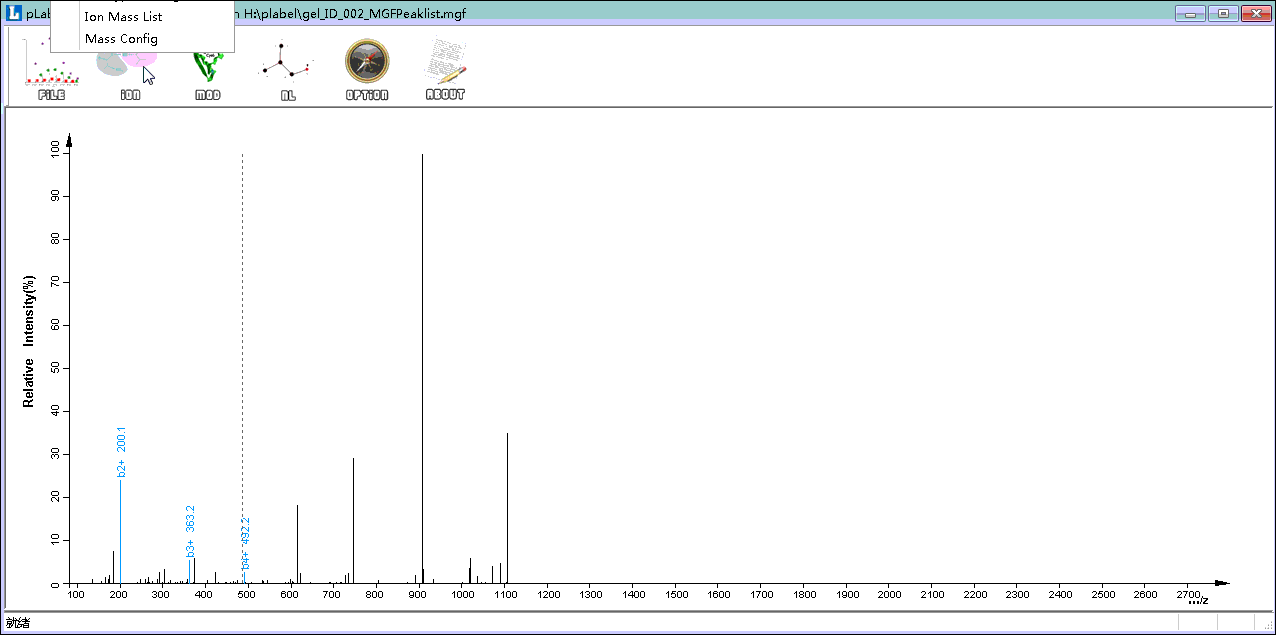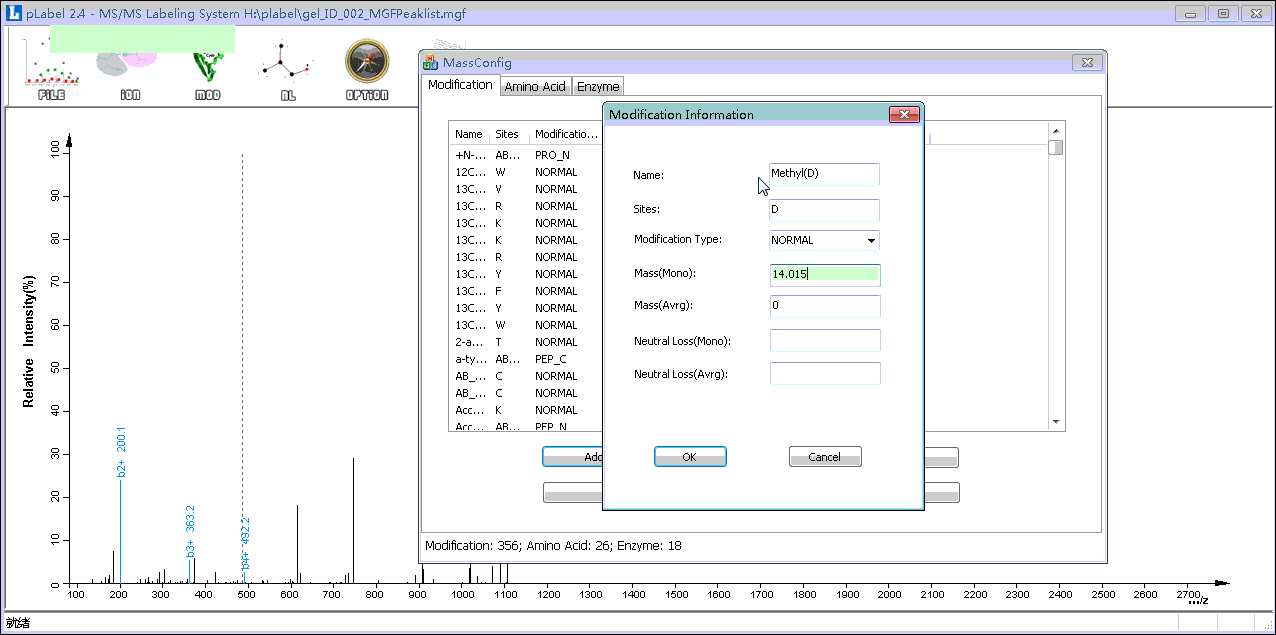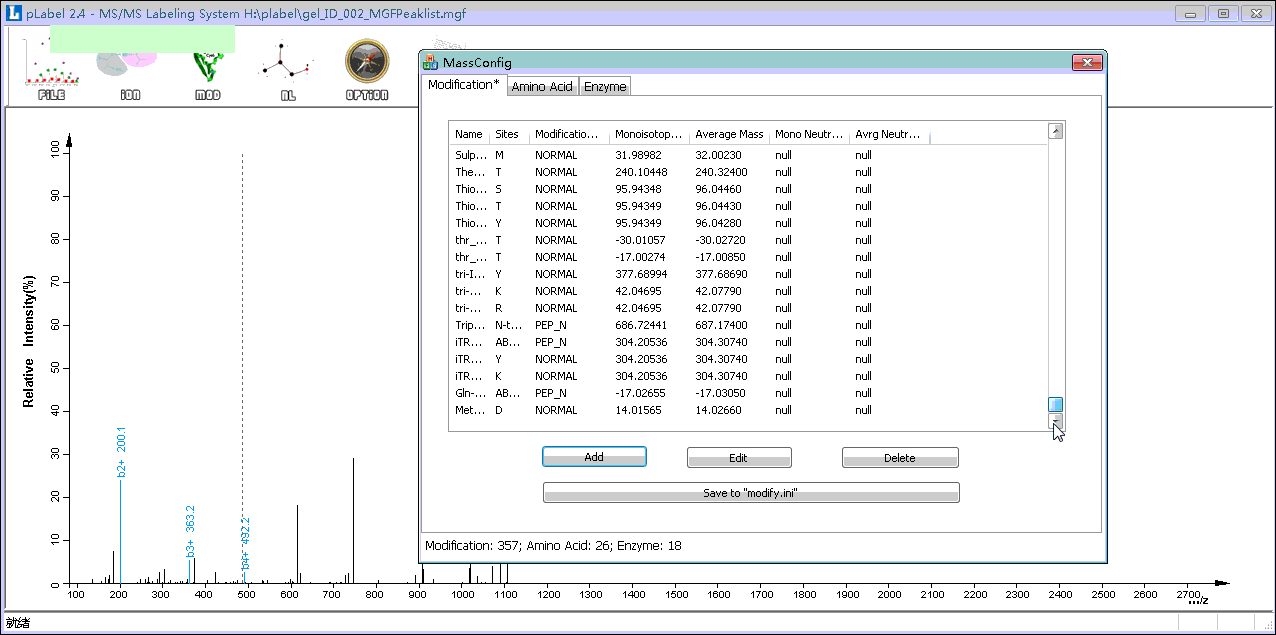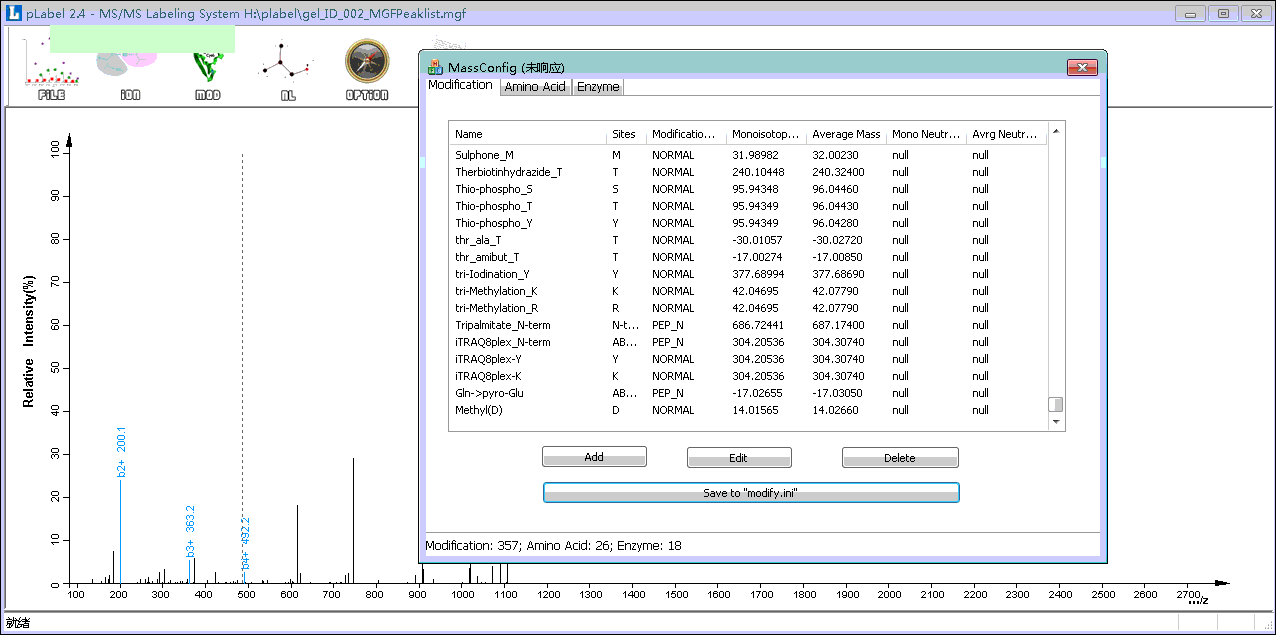
Step5. 添加未知修饰类型
我们发现,添加这两种修饰后,更多的碎片离子得到了标注(如图)
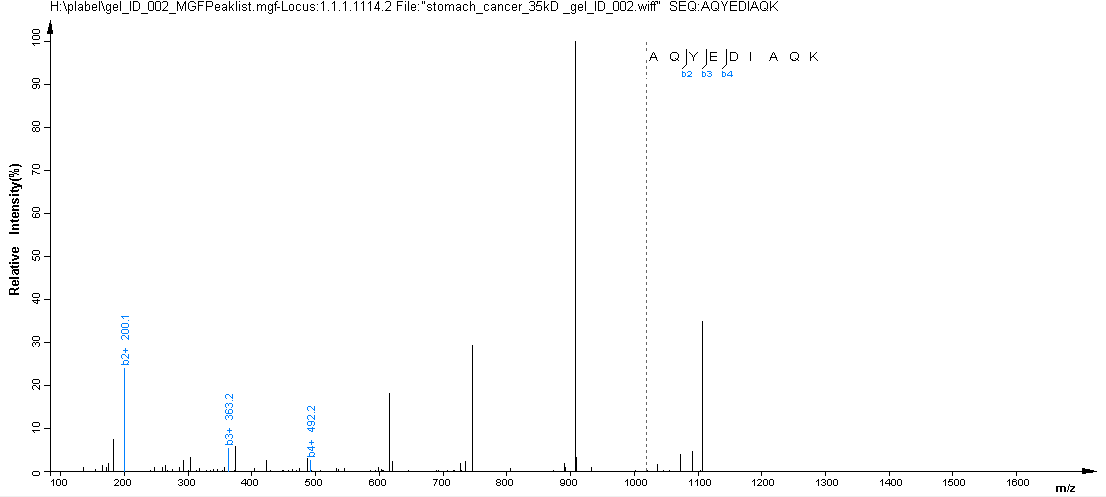
添加修饰前峰图
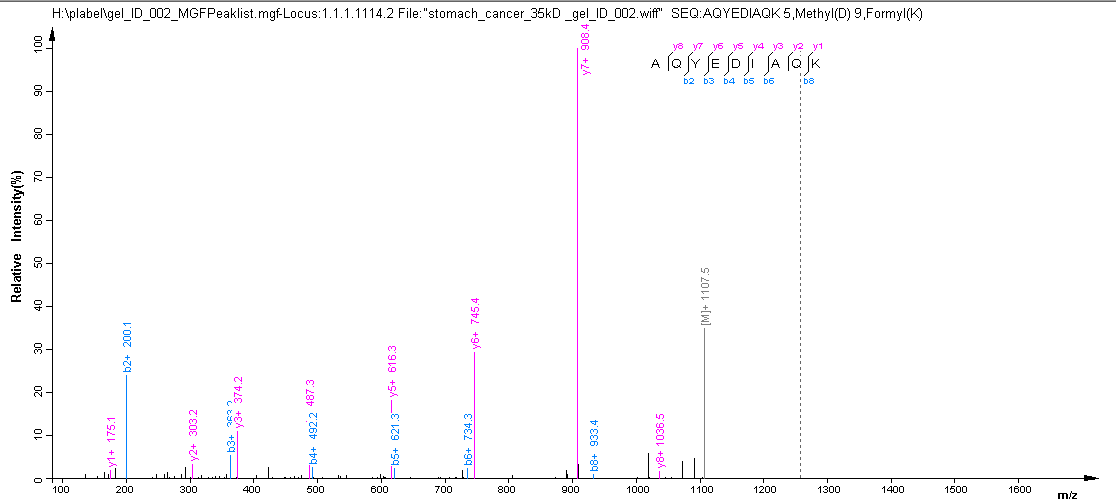
添加修饰后峰图
除了上述介绍的功能外,Plabel还可以自定义标峰的颜色、字体,以及需要显示的信息。童鞋们可以探索一下。学习过程有什么问题请留言哦!
上一篇:DNA测序基本原理及流程