技术专题
蛋白质组联合乙酰化修饰组学揭示玉米真菌诱导机制
文章来源:genecreate
作者:genecreate
发布时间:2018-10-24 15:56
题目:Fungal-induced protein hyperacetylation in maize identified by acetylome profiling
乙酰化修饰组学研究玉米真菌诱导乙酰化机制
期刊:PNAS
影响因子:9.504
主要技术:乙酰化蛋白质组,iTRAQ
研究背景
赖氨酸乙酰化是关键的翻译后修饰,可以调节一系列生物过程中多种蛋白质。组蛋白乙酰化在植物防御中发挥重要作用,并且已知编码乙酰转移酶的病原体效应蛋白可以直接乙酰化宿主蛋白。然而,尚不清楚内源性植物酶是否可以在免疫应答期间调节蛋白质乙酰化。效应分子HC-毒素(HCT),一种由真菌玉米圆斑病菌种1(C. carbonum race 1)产生的组蛋白去乙酰化酶抑制剂,可以增强病原体感染玉米植物的能力。
研究内容及结果
1. 玉米免疫应答定量分析
作者选择hm1A玉米突变株作为研究对象,该突变株编码缺陷NADPH依赖性还原酶(HCTR),不能使HCT失活,表现出对C. carbonum race 1的易感。随后,作者将突变株进行实验处理:(i)mock,模拟对照组;(ii)HCT,外源HCT处理组;(iii)Tox-,HCT缺陷型C. carbonum race 1感染组;(iv)Tox+,HCT表达型C. carbonum race 1感染组(图1A)。实验处理22h后收集组织样本(Tox +菌株易感染期间出现视觉症状之前)。然后,使用iTRAQ定量蛋白质组和乙酰化修饰蛋白质组技术进行质谱检测分析(图1B),在本次实验中,iTRAQ蛋白质组共定量到3636个蛋白,乙酰化修饰蛋白质组共定量到912个蛋白和2791个乙酰化修饰位点。
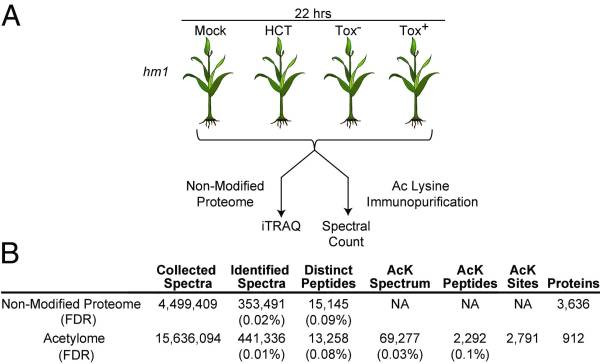
图1 实验设计及蛋白质组学定量结果
2. HCT诱导色氨酸生物合成蛋白和Bx植物抗毒素水平
为了研究HCT靶向促进C. carbonum毒力的特定生物过程,作者对获得的蛋白质组学结果进行深入分析,筛选关键蛋白,发现了171个上调蛋白和116个下调蛋白(图2A)。通过GO功能富集分析(图3),发现差异蛋白在吲哚/色氨酸生物合成途径发生显著富集,同时HCT组和Tox+组中吲哚/色氨酸生物合成中每一步所需的蛋白质表达均表现出上调趋势(图2B)。吲哚是Bx植物抗毒素前体物质,为了确定上调的蛋白水平是否引起Bx的积累,作者选择44小时后测量样本总Bx含量,发现HCT组和Tox +组处理Bx含量水平明显增加。结果表明C. carbonum 可以通过改变吲哚/色氨酸生物合成以实现感染玉米植株。
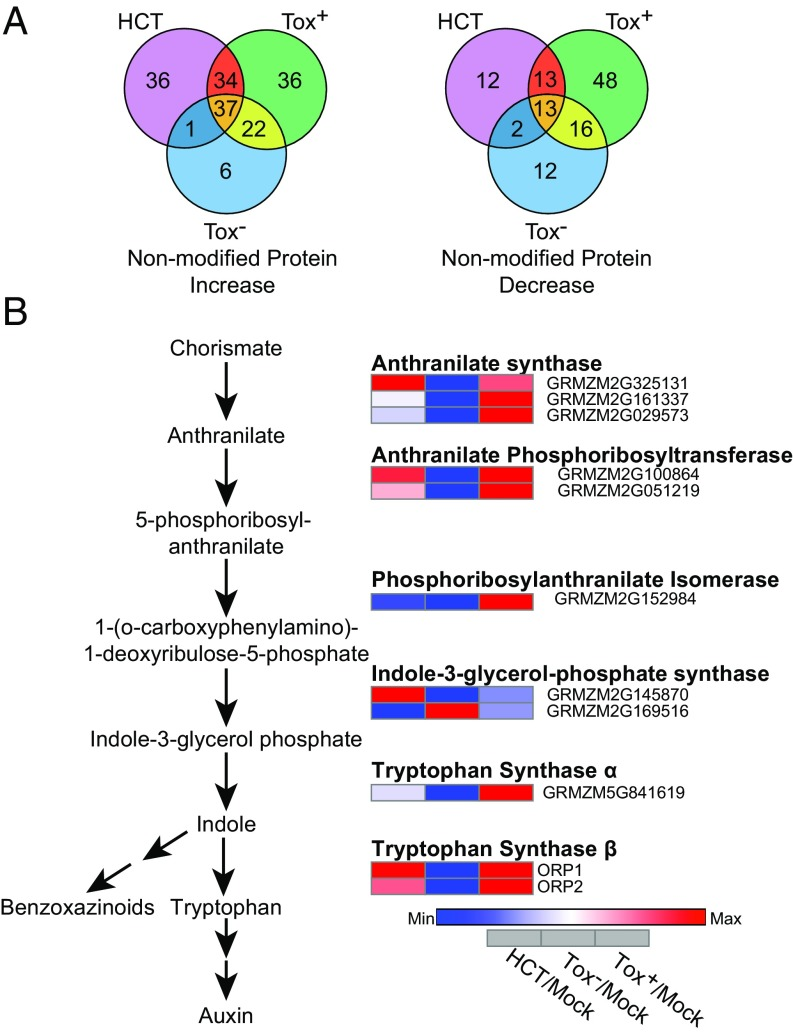
图2 吲哚/色氨酸合成途径中差异表达蛋白

图3 GO富集分析
3. 病原体感染改变组蛋白和非组蛋白乙酰化
根据乙酰化修饰组学结果,发现HCT处理后有62个乙酰化修饰肽段(155个乙酰化修饰位点)上调,9个乙酰化修饰肽段(12个乙酰化修饰位点)下调(图4D)。随后作者选用识别四乙酰化组蛋白H4(H4K5/ 8/12/16)的商业抗体进行WB验证实验,修饰组学和独立的WB结果均表明HCT和Tox+感染诱导的组蛋白H4四乙酰化发生在组蛋白H4赖氨酸残基5、8、12和16上(图4A,B)。此外,对乙酰化修饰GO注释结果统计发现,HCT组中31个、Tox+组中有8个都和转录调控相关(图4E),这些超乙酰化转录调节蛋白包括基因特异性转录因、一般转录因子TFIID(TAF5和TAF6)的亚基、转录辅阻遏物、染色质重塑酶和HAT酶等。已有研究结果表明,这些超乙酰化转录调节蛋白能够在植物免疫中发挥作用。
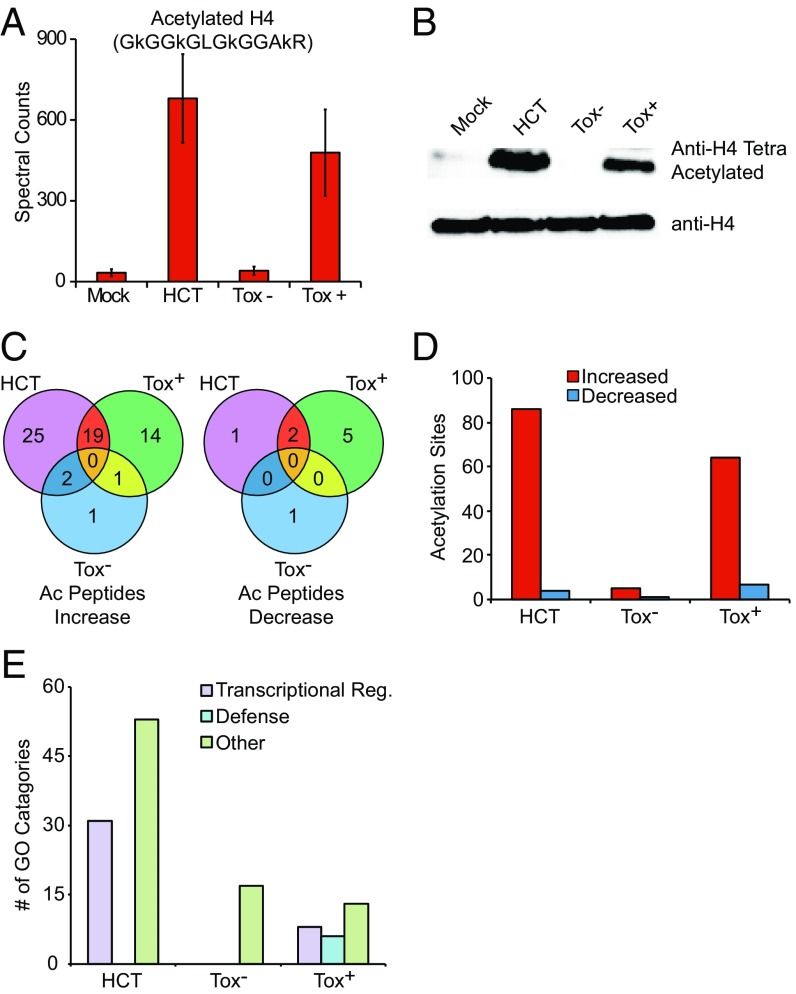
图4 乙酰化修饰蛋白质组学分析
为了深入研究玉米整体乙酰化修饰组成,作者使用MapMan分析了乙酰化蛋白质的功能类别分布。结果显示35个主要MapMan区域中有32个功能类别中的蛋白质含有乙酰化蛋白质(图5),这说明HCT可以广泛调控玉米中蛋白乙酰化修饰。综上,HCT不仅可以调控组蛋白的乙酰化修饰,还可以调控非组蛋白的乙酰化修饰。

图5 MapMan分析
文章小结
在玉米中存在广泛的乙酰化修饰,利用外源HCT处理玉米hm1A突变株,发现HCT能够通过调节植物酶活性进而改变免疫应答过程中的组蛋白和非组蛋白乙酰化水平,而病原菌在感染玉米植株时,可能利用HCT重编程植物中与感染相关基因的转录,导致植物产生无效的防御反应。
解析文献
Justin W. Walley, Maxwell R, et al. McReynolds Fungal-induced protein hyperacetylation in maize identified by acetylome profiling. PNAS, 2018 , 115 (1) :210-215
参考文献
1. Liu H, Sadygov RG, Yates JR. A model for random sampling and estimation of relative protein abundance in shotgun proteomics. Anal Chem, 2004, 76:4193–4201.
2. Smith-Hammond CL, Swatek KN, et al. Initial description of the developing soybean seed protein Lys-N(e)-acetylome. J Proteomics, 2014, 96:56–66.
3. Downey M, et al. Acetylome profiling reveals overlap in the regulation of diverse processes by sirtuins, gcn5, and esa1. Mol Cell Proteomics, 2015, 14:162–176.
4. Xiong Y, Peng X, Cheng Z, Liu W, Wang G-L.A comprehensive catalog of the lysine-acetylation targets in rice (Oryza sativa) based on proteomic analyses.J Proteomics, 2016, 138:20–29.
上一篇:代谢组学分析流程及常用软件