技术专题
调节T细胞的蛋白质组学研究揭示其在保护细胞信号通路中的适应性
文章来源:genecreate
作者:genecreate
发布时间:2018-07-25 10:29
题目:Proteomic Analyses of Human Regulatory T Cells Reveal Adaptations in Signaling Pathways that Protect Cellular Identity(人调节T细胞的蛋白质组学研究揭示其在保护细胞信号通路中的适应性)
期刊:Immunity
影响因子:19.734
主要技术:转录组、蛋白质组
研究背景
Treg cells(调节性T细胞)因其转录因子FOXP3的表达和抑制免疫应答的能力构成了独特的CD4+T细胞谱系。Treg 细胞可以保护免疫耐受,抑制免疫细胞造成的组织损伤,促进组织修复。Treg细胞也有不好的一面,它可以阻碍机体对癌细胞的免疫能力,有研究认为Treg这部分的功能缺失可能是造成人类自身免疫疾病和过敏发生的基础。因此,需要有针对性的促进或抑制人类疾病中的Treg细胞功能。Treg细胞和Tconv细胞密切相关,作者希望能够在分子水平上对二者进行详细研究和区分。
研究内容及结果
1. Treg细胞蛋白表达特征
作者从健康人供体的外周血单核细胞(PBMCs)分离CD4 + T细胞亚群。这些细胞亚群包含nTconv细胞(CD45RA + CD25-)、记忆(m)Tconv细胞(CD45RA-CD25-)、nTreg细胞(CD45RA+CD25hi)和eTreg细胞(CD45RA-CD25hi)(图1A)。两种Treg细胞亚群都表达FOXP3和Helios并且缺乏IL-7受体α链(CD127),而nTconv和mTconv细胞亚群则相反,它们缺乏FOXP3和Helios,但是能表达CD127。使用高分辨率质谱(MS),作者鉴定了平均35,744±3,757个肽段,5,955±344个蛋白gurup。在所有五个CD4+T细胞亚群中定量了4,358种不同的蛋白质。其中,422种蛋白质基于其非标记定量(LFQ)值在CD4+T细胞亚群中表现出差异表达(FDR <0.05)。 总数据集的主成分分析(PCA,图1B)、Pearson相关分析(图1C)、差异表达蛋白的层次聚类(图1D)证实了生物重复的密切相关性。作者分离CD4+T细胞亚群的原则并不是基于细胞谱系,而是根据CD45RA的表达(图1D)。富集分析显示,幼稚Treg和Tconv细胞共享参与磷酸戊糖代谢、NADP代谢和染色质组织等过程中的过表达蛋白(图1D),而eTreg和mTconv细胞共享参与蛋白质合成、细胞运输、信号传导和凋亡的过表达蛋白(图1D)。基于分化阶段蛋白质表达的相似性可部分反映淋巴组织(幼稚细胞)与非淋巴组织(效应细胞)的优先定位。从聚类结果可以发现具体的Treg细胞蛋白特征,如nTreg和eTreg细胞共享某些高表达蛋白(cluster1)和低表达蛋白(cluster9-10),而eTreg细胞则独自表现出在cluster2中蛋白高表达以及在cluster6中蛋白低表达。
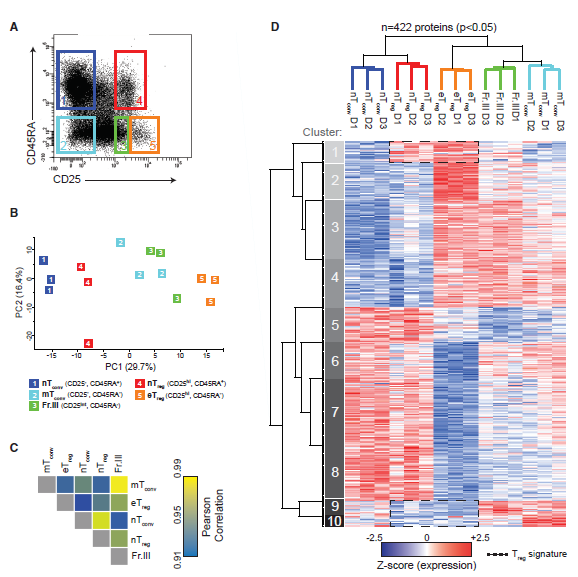
图1 CD4+T细胞亚群蛋白组学结果
2. Treg细胞的mRNA表达特征
Treg细胞的蛋白组学结果与已发表的相似的细胞亚群的转录组学结果关联性较低。为了进行直接比较,作者对五个CD4+T细胞亚群进行了全基因组mRNA深度测序。也基于它们的转录组分析发现,nTreg和nTconv细胞聚集在一起并远离三种效应和记忆表型CD4+T细胞群(图2A)。在五个CD4 + T细胞亚群(图2B)之间总共649个差异表达mRNA(p <0.05),包括预期的标志基因如IL-7R、IKZF2(HELIOS)和效应细胞因子。作者分析发现,eTreg细胞的mRNA表达特征既有特异性(图2B,cluster7;),也有和其他Treg细胞的一致的共性,如都可以表达FOXP3、IL2RA、TIGIT等分子(图2B和2C,cluster6)。已经发表的文章结果也证明了作者的发现。作者发现,Treg细胞蛋白质组学结果和转录组结果中只有三个分子(FOXP3,SHMT2和SWAP70)表达量趋势上发生重叠(图2F)。事实上,553 个mRNA和409个蛋白质在五个CD4+ T细胞亚群之间显着差异表达,并且可以在两个水平上定量,但是二者仅重叠48个(图2G)。因此,与众多报道的文献结果一致,相对于转录组结果,蛋白质组学会获得明显不同的结果(稳定状态下分析的细胞中)。
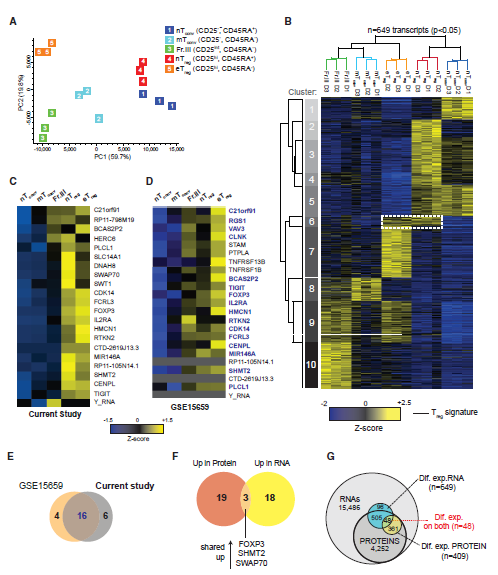
图2 CD4+ T细胞亚群mRNA表达结果
3. 蛋白质水平与mRNa水平的区别
为了定量比较蛋白质和mRNA水平,作者使用基于强度的绝对定量分析蛋白质数据(iBAQ)。发现蛋白质和mRNA的丰度水平都超过五个数量级(图3A和3D)。在蛋白水平中,丰度最高的是核糖体和代谢蛋白(图3B),在mRNA水平中,丰度最高的是编码参与(免疫)细胞、信号传导和功能的核糖体组分和分子(图3E),差异蛋白的丰度通常要比mRNA要高。
作者对4792个mRNA-protein对的表达量在转录组和蛋白组水平上进行定位,发现其相关系数约为0.44±0.01。422种差异表达的蛋白质中,在mRNA水平检测到409种,并进行定量比较(图3G)。虽然蛋白质和mRNA表达水平通常相关(例如,FOXP3I、KZF2),但是大多数仅有一种(蛋白或mRNA)的表达量达到差异统计标准,这就解释了差异表达的蛋白质和mRNA之间的有限重叠(图2F)。作者发现一些分子仅在mRNA或蛋白质水平上真正差异表达(图3H),这说明细胞调节具有一定的层次范围。例如,对于FTH1(图3G),是一个已知在严格的翻译控制下的蛋白质(Hentze等人,2010),它在nTreg和eTreg细胞中丰度都很高;STAM2在mTconv细胞中虽然mRNA水平低,但是蛋白丰度很高(图3G), 另一方面,在nTreg和eTreg细胞中,mRNA上高表达但蛋白质水平并不高(图3G)。
作者认为联合蛋白质组和转录组分析可以增加对通路分析的可信度。这样的组合数据有力地论证了与其他CD4+ T细胞亚群相比,核因子kB(NF-kB)和JAK-STAT途径在eTreg细胞中更容易脱敏(图3I)。总之,其研究结果强调了蛋白质组学分析对细胞类型功能表征的重要性。
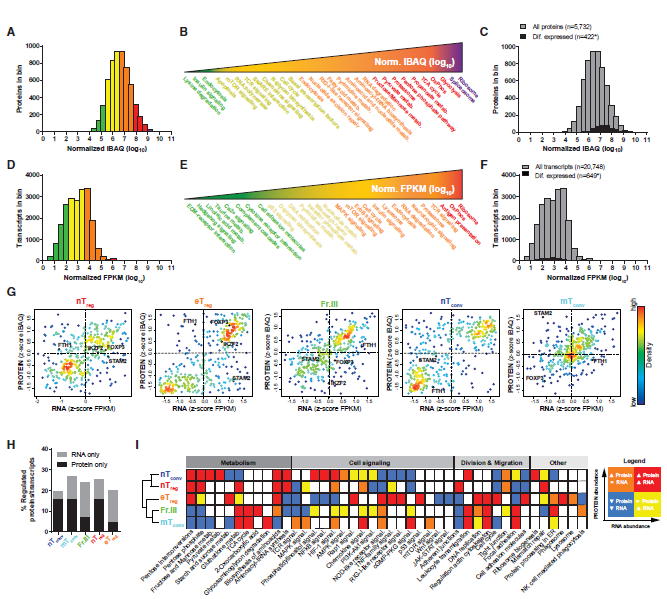
图3 mRNA和蛋白表达的通路富集分析结果
4. Treg细胞具有稳定的蛋白质特征
作者定义的常见Treg细胞特征由22个具有较高表达的蛋白质和29个低表达蛋白质组成(图4A)。 免疫印迹和流式细胞术证实了这些蛋白质中的8种蛋白质组学数据。 在早期的Treg细胞蛋白质组学研究中,多是基于CD4+CD127-CD25+和CD4+CD127+ CD25-细胞,覆盖的蛋白质组较少,所以并没有找到类似作者发现的组学特征。常见的Treg细胞特征包括FOXP3、IKZF2(Helios)、代谢蛋白GK、UGP2和SHMT2、铁储存蛋白铁蛋白重链和轻链(FTH1,FLT),以及溶酶体蛋白ASAH1、GGH、GUSB、SGSH和PLBD2,与Tconv细胞相比,它们都在Treg中以高丰度表达(图4A和4B)。 该特征还包括糖酵解酶HK1、ME2、脂肪酸氧化酶AP00和线粒体脂肪酸转运蛋白(CPT1A),与Tconv细胞相比,均在Treg中以低丰度表达。作者发现许多信号分子在常见的Treg细胞特征中是差异表达的,如脱氢泛素酶OTULIN的高表达,TNFα诱导的NF-kB活化的抑制剂和TNFRSF1A接头TRADD的低表达(图4A和4B),以及mRNA水平的TNFRSF1B的高表达,表明Treg细胞在TNFR信号传导中表现出适应性。脂质磷酸酶INPP5D(SHIP-1)的高表达抑制PI3K-AKT信号传导,mTOR活化剂RPS6KA1和RPS6KA3的低表达,同样也表明PI3K / AKT / mTOR途径的适应性,Treg细胞中STAT4和NFATc2的低表达突出。
为了确定上文鉴定的蛋白质表达模式的稳定性,作者在体外T细胞扩增后进行蛋白质组学分析。通过CD3、CD28和IL-2刺激将nTreg和nTconv细胞在体外扩增2周,孵育4天,并通过FOXP3、Helios、CTLA-4和CD25染色以及FOXP3基因TSDR甲基化验证它们的身份。值得注意的是,即使在通过CD3和CD28途径重新激活后,Treg细胞核心特征中大多数蛋白质特异性表达模式也基本上是保守的(图4C,4D)。作者认为Treg细胞在某些代谢功能以及重要信号通路的组成中与Tconv细胞本质上不同。
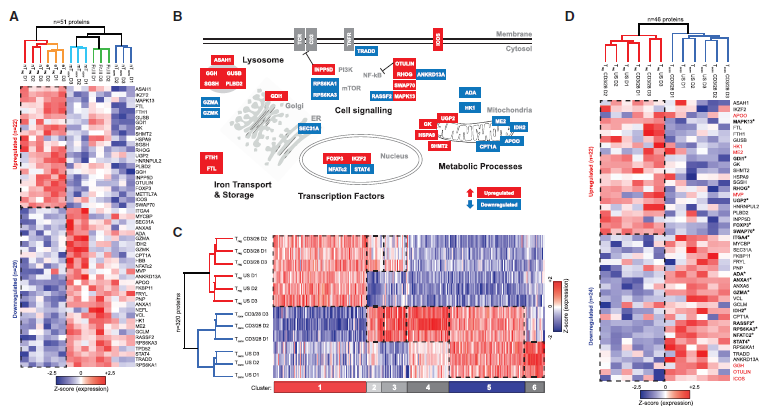
图4 常见的Treg细胞蛋白组学特征
5. eTreg细胞特有的蛋白组学特征
除了常见的Treg细胞特征外,作者发现eTreg细胞与其他CD4+ T细胞亚群相比有独特的蛋白质簇表达特征:具有相对高的(eTreghi)和低的(eTreglo)表达(图5A)。eTreghi簇包括参与DNA复制(MCM2,-3,-4,-6,-7,FEN1)、有丝分裂(CORO1C,TUBA1B,TUBB,MYH9)(图5A和5B)和细胞凋亡(FAS,CASP3)的蛋白质,这和已有研究一致,说明这些细胞正在发生分裂。在eTreghi簇中的41种蛋白质中,17种在体外Treg细胞扩增后仍显示出上升趋势,其中5种达到统计学显着性。同样,eTreglo簇中39种蛋白中的25种在体外扩增的Treg细胞中保持低表达。eTreglo簇包含多种调节细胞凋亡敏感性的GIMAP(图5A和5B)。重要的是,eTreglo簇中几乎40%的蛋白质在细胞信号传导中具有功能,它们包括NF-kB途径的多种成分(PRKCB、DPP4、NF-kB1、NF-kB2)、细胞因子受体途径(IL-7R、INPP4B、STAM2、STAT3)和TCR途径(TRAT1、VAV1、THEMIS)。即使在体外培养之后,Treg细胞中许多蛋白质的表达相对较低,表明这种对信号通路的明显脱敏作用,并不仅仅是通过这些通路在体内激活信号的负面反馈调节。
FOXP3可以与关键转录因子(如IL17A和IL4)发生物理相互作用并且具有可以淬灭它们反式激活Tconv细胞效应基因的能力。反之亦然,如YY1的因子可以抑制FOXP3介导的Treg细胞程序的控制。作者为了确定Treg细胞中FOXP3的相对浓度是否足以“压倒”这样的相互作用因子,使用蛋白质组学标尺方法确定了每个细胞的蛋白质拷贝数,发现FOXP3在eTreg细胞中的拷贝数超过其许多伴侣蛋白,其中差别最大的是转录因子,如NFATc1和STAT4(图5 C)。然而,对于一些转录因子(YY1、RUNX1和NFATc2),FOXP3过量仅为2至3倍,这可以解释为什么FOXP3的适度减少允许Treg细胞产生效应细胞因子。最后,作者发现mTconv细胞中FOXP3的表达水平可能不足以有效中和其伴侣转录因子(图5C)。
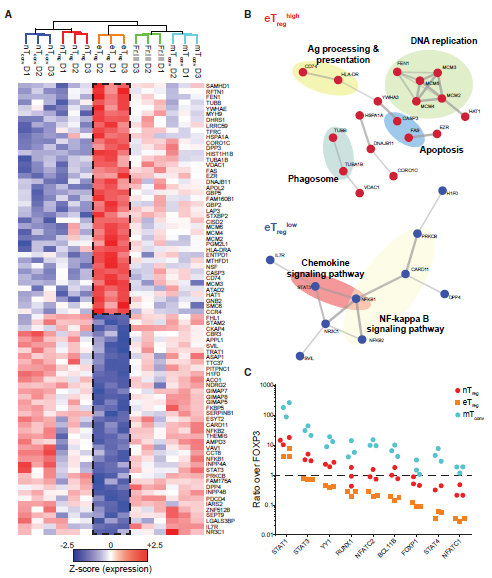
图5 eTreg细胞蛋白特征
6. eTreg细胞具有效应基因表达的迟钝途径
作者发现与Tconv细胞相比,eTreg细胞的IPA分析显示TCR、模式识别受体(PRR)、细胞因子受体和TNFRSF家族诱导的NF-κB途径信号传导具有适应性。此外,通过NF-kB1和NF的流式细胞术证实,eTreg细胞中NF-kB1(p50)、NF-kB2(p52)和RELA(p65)的mRNA水平较低。因此,在eTreg细胞中,NF-kB1的反cd3和反cd28诱发的核核易位被削弱了,但在nTreg细胞中却没有。NFATc1和NFATc2蛋白水平在nTreg细胞和eTreg细胞中都较低。此外,NFATc2在普通Treg细胞的蛋白中含量较低,在Treg细胞在体外(图4A和4D)后保持低表达。最后,在eTreg细胞转录组(图6A)中,带有NFATc2绑定元素的基因含量降低(图6A),这表明该因素在体内的这些细胞中不那么活跃。
炎性细胞因子控制Treg细胞行为可能会对其稳定性造成影响,在传递细胞因子受体信号的STAT转录因子中,STAT3、STAT6,尤其是STAT4在eTreg细胞中表现出低表达。STAT4表达在mRNA和蛋白质水平均较低,STAT4的低蛋白质表达是常见Treg细胞蛋白特征的一部分。在eTreg细胞转录组中,带有STAT4结合元素的基因也没有得到充分的表达(图6A),这支持了体内eTreg细胞中STAT4活性降低的观点。通过响应IL-12和I型IFN磷酸化Y693,在人CD4 + T细胞中激活STAT4,后者在人CD4 + Tconv细胞中诱导最强的早期STAT4磷酸化。这些细胞因子一致地诱导Treg细胞中STAT4的磷酸化比从血液中新鲜分离的Tconv细胞中更少(图6C),并且这种差异在体外扩增的细胞中甚至更为显着(图6E)。STAT4是IFNg基因表达的主要调节因子,因此,作者推断STAT4的低表达可能使Treg细胞与炎性细胞因子诱导IFN-g隔离。与此假设一致,IFN-α并且IL-12不能在Treg细胞中诱导IFN-g产生(图6F)。然而,当故意过表达STAT4时,这些细胞因子确实激发了IFN-g的产生Treg细胞(图6G)。值得注意的是,尤其是当用IFN-α或IL-12刺激Treg细胞时,STAT4的过表达也诱导了IL-2的产生(图6G)和FOXP3表达的显着丧失(图6H和6I)。这些发现表明STAT4的低表达有助于在炎性环境中保护Treg细胞。
因为Treg细胞需要来自炎性细胞因子的输入,所以虽然STAT4表达低,但是作者仍验证了Treg细胞是否能够对I型IFN起反应。结果发现,在Treg和Tconv细胞中均等地诱导STAT1磷酸化(图6B和6D)。 此外,IFN-α容易在抗CD3和抗CD28活化的Treg细胞中诱导转录因子T-bet和趋化因子受体CXCR3的表达(图6J,6K)。 总之,这些发现表明STAT4的选择性低表达允许Treg细胞响应炎性细胞因子(例如归巢至发炎组织),而不损害Treg细胞。

图6 Treg细胞中选择性缺乏STAT4激活
7. 在Fr.III细胞中可以区分不同的细胞群
Fr.III细胞是CD4+CD25+并且表达FOXP3,可以产生炎性细胞因子并且在体外缺乏强大抑制能力的细胞。该群体中的许多细胞表达CD127表明它是含有类似Treg或Tconv细胞的细胞的混合群体。为了更好地表征这一群体,作者根据CD127是否表达将其分成两个部分,然后进行每个级分的蛋白质组学分析,并与nTconv、mTconv、nTreg和eTreg细胞的蛋白质组一起进行分析。层次聚类分析和PCA显示CD127+ 亚群与mTconv细胞密切相关(图7A和7B),说明CD127用于区分Tconv和Treg细胞有用性。值得注意的是,CD127- 亚群与eTreg细胞几乎无法区分。CD127- Fr.III和eTreg细胞之间的紧密蛋白质组关系表明前者可能是真正的Treg细胞群,然而,FOXP3中的TSDR在CD127- Fr.III细胞中比在eTreg细胞中更加甲基化(图7C)。此外,CD127-Fr.III细胞含有大部分产生一种或多种效应细胞因子的细胞(图7F),这与eTreg细胞不同,因为eTreg细胞大多缺乏这种能力。
并非CD127- Fr.III群体中的所有细胞都产生效应细胞因子,也并非eTreg细胞群中的所有细胞都缺乏产生此类细胞因子的能力,这表明即使这些明确定义的细胞群体仍可能是异质的。因此,作者在蛋白质组数据集中搜索了可能有助于区分具有不同功能特性的细胞的标记物。之前已有研究报道,在Treg细胞上发现的两种标记物与阳性(CD49d)或阴性(CCR4)标志物与群体产生效应细胞因子的相对能力相关(图7D)。流式细胞术分析显示这些标志物表现出互斥的表达模式(图7E),eTreg细胞群中产生IL-2或IL-17的少数细胞在表达CD49d的细胞中最为突出,这和已有的报道结果一致。然而,仅CD49d-CCR4 +表型完全排除了完全产生效应细胞因子的eTreg细胞(图7G)。在CD127-Fr.III群体中观察到类似的趋势,其中CD49d-CCR4 +群体中不存在产生IL-17和IFN-g的细胞。然而,该群体在产生IL-2的能力方面仍然不同于相应的CD49d-CCR4 + eTreg细胞亚群(图7G)。FOXP3的相对蛋白水平与每个群体中产生细胞因子的细胞的比例成反比。因此,FOXP3表达逐渐从eTreg细胞(最高)降低至Fr.III CD127-和Fr.III CD127 +(最低,但仍然高于mTconv细胞)。此外,在每个群体中,CCR4+CD49d-细胞总是表达比CCR4-CD49d +细胞更高的FOXP3。最后,TSDR仅在eTreg细胞中完全去甲基化,并且主要在CD127-CCR4+CD49d-Fr.III细胞中甲基化,其仅产生IL-2,但不产生其他炎性细胞因子。
因此,作者的蛋白质组学数据使其发现CCR4和CD49d区分具有不同能力的细胞,以在eTreg细胞和Fr.III群体内产生效应细胞因子。这些标记物与常用的Treg细胞标记物组合的组合可用于细胞纯化和诊断目的。例如,它们有助于阐明肿瘤中FOXP3+ CD4+T细胞的存在与患者愈后之间的关联,特别是对于癌症如结肠癌等。

图7 蛋白质组学揭示了Fr.iii和eTreg细胞之间的功能异质性
文章小结
通过对人类调节和常规CD4+T(Tconv)细胞的各种群体进行蛋白质组学和转录组学,获得调节性T(Treg)细胞特性的分子特征,鉴定并定义了所有Treg细胞的蛋白质表达特征,以及定义效应Treg细胞的独特特征。发现了Treg细胞中的代谢特征,以及细胞因子、TCR和共刺激受体信号传导途径的特异性适应性:适应——选择性STAT4缺陷——保护Treg细胞稳态模型,该途径通过炎性细胞因子起作用,而这些信号仍然可以通过其他途径诱发关键的转录因子和引导受体。此外,作者的研究揭示了识别具有不同功能特性的FOXP3+ CD4+T细胞的表面标志物。作者的研究结果表明,信号通路中的适应性保护Treg细胞,并为进一步研究Treg细胞生物学提供了资源。
解析文献
Eloy Cuadrado, Maartje van den Biggelaar, et al. Proteomic Analyses of Human Regulatory T Cells Reveal Adaptations in Signaling Pathways that Protect Cellular Identity. Immunity, 2018.
参考文献
1. Vogel, C., and Marcotte, E.M. (2012). Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat. Rev. Genet. 13, 227–232.
2. Arpaia, N., Green, J.A., et al . (2015). A distinct function of regulatory T cells in tissue protection. Cell 162, 1078–1089.
3. De Rosa, V., Galgani, M., et al. (2015). Glycolysis controls the induction of human regulatory T cells by modulating the expression of FOXP3 exon 2 splicing variants. Nat. Immunol16, 1174–1184.
4. Oh, H., Grinberg-Bleyer, Y., et al. (2017). An NF-kB transcription-factor-dependent lineage-specific transcriptional program promotes regulatory T cell identity and function. Immunity 47, 450–465.e5.
上一篇:蛋白质组学到底是研究什么