技术专题
基因定点突变全攻略
文章来源:原创
作者:genecreate
发布时间:2022-11-01 10:41
一、定点突变的目的
把目的基因上面的一个碱基换成另外一个碱基。
二、定点突变的原理
定点突变是指通过聚合酶链式反应(PCR)等方法向目的DNA片段(可以是基因组,也可以是质粒)中引入所需变化(通常是表征有利方向的变化),包括碱基的添加、删除、点突变等。定点突变能迅速、高效的提高DNA所表达的目的蛋白的性状及表征,是基因研究工作中一种非常有用的手段。
体外定点突变技术是研究蛋白质结构和功能之间的复杂关系的有力工具,也是实验室中改造/优化基因常用的手段。蛋白质的结构决定其功能,二者之间的关系是蛋白质组研究的重点之一。对某个已知基因的特定碱基进行定点改变、缺失或者插入,可以改变对应的氨基酸序列和蛋白质结构,对突变基因的表达产物进行研究有助于人类了解蛋白质结构和功能的关系,探讨蛋白质的结构/结构域。而利用定点突变技术改造基因:比如野生型的绿色荧光蛋白(wtGFP)是在紫外光激发下能够发出微弱的绿色荧光,经过对其发光结构域的特定氨基酸定点改造,现在的GFP能在可见光的波长范围被激发(吸收区红移),而且发光强度比原来强上百倍,甚至还出现了黄色荧光蛋白,蓝色荧光蛋白等等。定点突变技术的潜在应用领域很广,比如研究蛋白质相互作用位点的结构、改造酶的不同活性或者动力学特性,改造启动子或者DNA作用元件,提高蛋白的抗原性或者是稳定性、活性、研究蛋白的晶体结构,以及药物研发、基因治疗等等方面。
通过设计引物,并利用PCR将模板扩增出来,然后去掉模板,剩下来的就是我们的PCR产物,在PCR产物上就已经把这个点变过来了,然后再转化,筛选阳性克隆,再测序确定就行了。
三、引物设计原则
引物设计的一般原则不再重复。
突变引物设计的特殊原则:
(1)通常引物长度为25~45 bp,我们建议引物长度为30~35 bp。一般都是以要突变的碱基为中心,加上两边的一段序列,两边长度至少为11-12 bp。若两边引物太短了,很可能会造成突变实验失败,因为引物至少要11-12个bp才能与模板搭上,而这种突变PCR要求两边都能与引物搭上,所以两边最好各设至少12个bp,并且合成多一条反向互补的引物。
(2)如果设定的引物长度为30 bp,接下来需要计算引物的Tm值,看是否达到78℃(GC含量应大于40%)。
(3)如果Tm值低于78℃,则适当改变引物的长度以使其Tm值达到78℃(GC含量应大于40%)。
(4)设计上下游引物时确保突变点在引物的中央位置。
(5)最好使用经过纯化的引物。
Tm值计算公式:Tm=0.41×(% of GC)–675/L+81.5
注:L:引物碱基数;% of GC:引物GC含量。
四、引物设计实例
以GCG→ACG为例:
5’-CCTCCTTCAGTATGTAGGCGACTTACTTATTGCGG-3’
(1)首先设计30 bp长的上下游引物,并将A (T)设计在引物的中央位置。
Primer #1: 5’-CCTTCAGTATGTAGACGACTTACTTATTGC-3’
Primer #2: 5’-GCAATAAGTAAGTCGTCTACATACTGAAGG-3’
(2)引物GC含量为40%,L为30,将这两个数值带入Tm值计算公式,得到其Tm=75.5(Tm=0.41×40-675/30+81.5)。通过计算可以看出其Tm低于78℃,这样的引物是不合适的,所以必须调整引物长度。
(3)重新调整引物长度。
Primer #1: 5’-CCTCCTTCAGTATGTAGACGACTTACTTATTGCGG-3’
Primer #2: 5’-CCGCAATAAGTAAGTCGTCTACATACTGAAGGAGG-3’
在引物两端加5mer(斜体下划线处),这样引物的GC含量为45.7%,L值为35,将这两个数值带入Tm值计算公式,得到其Tm为80.952(Tm=0.41×47.5-675/35+81.5),这样的引物就可以用于突变实验了。
五、突变所用聚合酶及Buffer
引物和质粒都准备好后,当然就是做PCR喽,不过对于PCR的酶和buffer,不能用平时的,我们做PCR把整个质粒扩出来,延伸长度达到几个K,所以要用那些GC buffer或扩增长片段的buffer,另外,要用保真性能较好的PFU酶来扩增,防止引进新的突变。
除了使用基因定点突变试剂盒,如Stratagene和塞百盛的试剂盒,但价格昂贵。可以使用高保真的聚合酶,如博大泰克的金牌快速taq酶、Takara的PrimeSTARTM HS DNA polymerase。
六、如何去掉PCR产物
最简单的方法就是用DpnI酶,DpnI能够识别甲基化位点并将其酶切,我们用的模板一般都是双链超螺旋质粒,从大肠杆菌里提出来的质粒一般都被甲基化保护起来(除非你用的是甲基化缺陷型的菌株),而PCR产物都是没有甲基化的,所以DpnI酶能够特异性地切割模板(质粒)而不会影响PCR产物,从而去掉模板留下PCR产物,所以提质粒时那些菌株一定不能是甲基化缺陷株。
DpnI处理的时间最好长一点,最少一个小时吧,最好能有两三个小时,因为如果模板处理得不干净,哪怕只有那么一点点,模板直接在平板上长出来,就会导致实验失败。
七、如何拿到质粒
直接把通过DpnI处理的PCR产物拿去做转化就行了,然后再筛选出阳性克隆,并提出质粒,拿去测序,验证突变结果。
八、图示
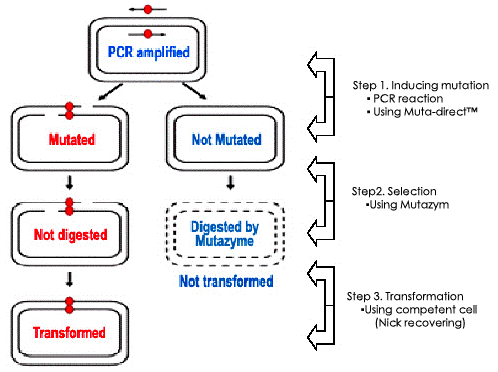
九、定点突变操作步骤
[A] 诱导突变基因(PCR反应)以待突变的质粒为模板,用设计的引物及Muta-direct™酶进行PCR扩增反应,诱导目的基因突变。
1. 设计点突变引物。
[注]参考引物设计指导
2. 准备模板质粒DN A
[注]用dam+型菌株(例如DH5α菌株)作为宿主菌。在end+型菌株中常有克隆数低的现象,但是对突变效率没有影响。提取质粒DNA时我们建议您使用本公司的质粒提纯试剂盒。
3. [选项]对照反应体系(50μl反应体系)
4. 样品反应体系(50μl反应体系)
5. PCR反应条件
[注]按如下参数设置PCR扩增条件。
6. PCR扩增反应完成后冰育5分钟,然后置于室温(避免反复冻融)。
[注] 按下列提供的PCR条件进行扩增,控制PCR循环数。注意当突变点位点超过4个时会发生突变率降低的现象。
[B] 突变质粒选择
PCR反应结束后使用Mutazyme™酶消化甲基化质粒从而选择突变质粒DNA。
1. 准备PCR反应产物
2. 加入1μl(10U/μl)Mutazyme™酶37℃温育1小时。
[注]当质粒DNA用量过多时Mutazyme™酶可能发生与样品反应不完全的现象。因此我们建议为了保证突变率请严格遵照实验步骤进行操作。如果突变率低,可以适当延长反应时间或增加Mutazyme™酶用量。
[C]转化
反应完毕后在质粒DNA上会产生缺口,当把这个质粒DNA转入E.coli中时请选择dam+型菌株,例如DH5α。
1. 将10μl样品加到50μl感受态细胞里,然后放置在冰上30分钟。
2. 接下来可以参照一般的转化步骤进行。
序列分析
通常当LB平板上出白色菌落则表明发生了突变。
为了证实这一结果,我们建议对白色单菌落进行测序分析。
先讲最简单的一个点的定点突变技术,其它较长片段的突变,删除,插入技术以后会慢慢奉上:在做实验之前,我们首先要搞清楚实验的目的和实验的原理。
实验的目的应该比较明确吧:就是要把自己的基因上面的一个碱基换成另外一个碱基。一般情况下我们会有几种可能使我们需要这样去做:
第一:我们吊出来的基因有点突变,相信这可能是大家经常会遇到的问题。基因好不容易吊出来,并装进了自己的载体,却发现有一两个碱基跟自己的预期序列或所有的公共数据库不匹配,然后暴昏。
大家实验室里面还是用Taq酶为主吧,Pfu这样的高保真酶大家应该用得不多吧,Taq酶的优点和缺点都很明显:优点就是扩增效能强,缺点就是保真性差,其错配机率是比较高的,相关数字忘了,大家可以去网上查那个数字,不过感觉如果是2000bp的基因,如果扩四五十个循环的话,很大机率会出现点突变,当然这也跟具体PCR体系里的Buffer有很大关系,详细情况这里就不讨论了。
第二:要研究基因的功能,在基因上自己选定位置更换碱基的保守序列,或者改造成不同的亚型,总之就是要人工改造碱基序列符合自己的实验需要,相信这也是那些研究基因的人经常的一种思路吧。
对于第一种情况:我们首先要分析出现碱基不匹配的位置是不是重要的位置,如果不是很重要,大可不必管它,比如说是三联密码子的最后一位,碱基的改变并没有引起相应氨基酸的改变,那么一般情况下也可以不去理它。另外,在NCBI上人类的基因的版本一直在变化,也就是说同一个基因有不同的版本,或者称不同的亚型,其碱基序列有些许的差异,只要自己克隆出来的碱基序列与其中一个相匹配,一般也就可以不做定点突变了。如果有时间没钱,那干脆重新PCR然后再克隆进自己的载体了,不过最好换个保真性好一点的酶如PFU,或者PCR循环数低一点,不过这些东西有时候也得靠运气啦。实在不行的话再来做定点突变。
对于第二种情况:这种情况下一般也就只能做定点突变了。
接下来开始聊一聊定点突变的原理吧,那个Stratagene试剂盒!上面有一个说明书,说得好像很正规,不过上面好多都是什么专利啊什么注意之类的话,看都不看,我们简明扼要地只讲实验方面,通过设计引物,并利用PCR将模板扩增出来,然后去掉模板,剩下来的就是我们的PCR产物,在PCR产物上就已经把这个点变过来了,然后再转化,筛选阳性克隆,再测序确定就行了。
大家马上就会想到几个问题了:
第一:引物怎么设计呢?
第二:模板怎么去掉呢?
第三:怎么拿到质粒呢?
对于第一个问题:怎么设计引物?
我只能讲一些原则,并举一些例子。
引物设计的原则其它贴子上都有讲,这里就不重复了:
不过这种突变引物要加上一个原则:
一般都是以要突变的碱基为中心,加上两边的一段序列,两边长度至少为11-12base pair。
若两边引物太短了,很可能会造成突变实验失败,大家应该都知道,引物至少要11-12个base pair才能与模板搭上,而这种突变PCR要求两边都能与引物搭上,所以两边最好各设至少12个base pair,并且合成多一条反向互补的引物。
这么说大家可能不是很清楚,那我就举个例子吧:
X71661.1 TATCAGGAGGAATTTGAGCACTTTCAACAAGAATTGGATAAAAAAAAAGAGGAATTCCAG 960
现有序列 TATCAGGAGGAATTTGAGCACTTTCAACAAGAATTGGATAAAAAAAAAGAGGAATTCCAG 924
*********************************************** ************
|----deletion
X71661.1 AAGGGCCACCCCGACCTCCAAGGGCAGCCTGCGGAGGAAATATTTGAGAGTGTAGGAGAT 1020
现有序列 AAGGGCCACCCCGACCTCCAAGGGCAGCCTGCGGAGGAAATATTTGAGAGTGTAGGAGAT 984(上面为目的序列,下面为现有序列:我们发现有一个A碱基的缺失,其直接结果是在表达蛋白时后面的氨基酸全部错配)
我们以它为中心设计引物:两边各至少12个碱基,左边由于含有较多的A造成引物GC%含量过低,故拉长引物使GC%含量不至过低,也使引物退火温度升高。
故合成引物CAACAAGAATTGGATAAAAAAAAAGAGGAATTCCAGAAG
并合成反向互补引物CTTCTGGAATTCCTCTTTTTTTTTATCCAATTCTTGTTG
其实也不一定要反向互补序列,只要反向引物也是两边都有大于12个碱基,同时符合引物设计的原则就行了。
引物合成公司有很多家,大家可以去寻找,不同厂家的引物在价钱质量上有一些差别,不过价钱一般都是一块多一个碱基,合成时间约为一周。
这样的结果是PCR时把整个质粒都给扩出来了,得到的PCR产物是一条链完整,另一链有缺刻的PCR产物
对于第二个问题:
怎么去掉模板呢?再简单的方法就是用DpnI酶,DpnI能够识别甲基化位点并将其酶切,我们用的模板一般都是双链超螺旋质粒,从大肠杆菌里提出来的质粒一般都被甲基化保护起来(除非你用的是甲基化缺陷型的菌株),而PCR产物都是没有甲基化的,所以DpnI酶能够特异性地切割模板(质粒)而不会影响PCR产物,从而去掉模板留下PCR产物,所以提质粒时那些菌株一定不能是甲基化缺陷株,不会那么凑巧吧,哈哈。
关于第三个问题:
直接把通过DpnI处理的PCR产物拿去做转化就行了,呵呵,然后再筛选出阳性克隆,并提出质粒,拿去测序(这个就不用我多说了吧),验证突变结果,一般都没问题的啦,我做了几十个突变了,到目前为止还没有做不出来的,呵呵,不要砸我啊。
下面讲一下具体的实验步骤以及一些实验中要注意的事情:
1、 根据现有基因设计引物;
2、 合成引物并准备好模板;
3、 PCR,
4、 DpnI处理酶切产物;
5、 转化酶切产物;
6、 筛选 阳性克隆;
7、 送测序并测全长。
最后就是庆祝啦,呵呵,没什么复杂的。
引物和质粒都准备好后,当然就是做PCR喽,不过对于PCR的酶和buffer,不能用平时的,我们做PCR把整个质粒扩出来,延伸长度达到几个K,所以要用那些GC buffer或扩增长片段的buffer,另外,要用保真性能较好的PFU酶来扩增,防止引进新的突变。
那种Quick change试剂盒分为几种不同的类型
什么QuikChange® Site-Directed Mutagenesis Kit标准点突变试剂盒 、QuikChange® XL Site-Directed Mutagenesis Kit长模板单点突变试剂盒(>8kb)从原理上是一样的,只是PCR的酶和BUFFER不一样,后面用了比较适合长片段扩增的酶和BUFFER罢了,没什么特别的东西。另外,DpnI处理的时间最好长一点,最少一个小时吧,最好能有两三个小时,因为如果模板处理得不干净,哪怕只有那么一点点,模板直接在平板上长出来,就会导致实验失败。
实验板长出来的菌有两种可能
一种是质粒DPNI没处理干净长出来的(模板),一种是PCR产物转化出来的
(突变体)
不过这两种菌长得一模一样^_^,即使提出质粒来也是一样(酶切和PCR都无法区分),除了测序,是分不出来的,
做PCR时也最好做一个负对照(不加引物),
实验管由于PCR时有引物,所以在DNPI处理前里面既含有模板又含有PCR产物,而对照管由于PCR时没放引物,所以在DPNI处理前里面只有模板。
如果两者都拿去DNPI处理
就能够证明模板已经被去除干净。
若实验顺利的话应该是:正对照长菌负对照不长菌。
如果出现正负对照都长菌,那么就是DpnI没处理好,
如果正负对照都不长菌,那么有两种可能,一种是PCR阴性,也就是说PCR出问题了,另外一个可能就是转化出问题了。要搞清楚是哪个问题,跑胶说明不了问题,那就做个转化的对照,拿试剂盒的对照实验去试感受态,马上就能知道转化有没问题。
如果正对照很多菌,负对照有几个菌,那么就是DPNI处理得不干净,这个时候就得靠运气了^_^
大家有什么问题我们可以继续讨论。
另外,如果大家既没有DpnI酶也没有好的PCR酶和BUFFER的话,那也有其它办法进行定点突变,只是麻烦一点,如果大家有需要的话,我会把方法贴上来。
对于多点突变技术及较长片段的缺失插入技术,同样的,如果大家有需要的话,我会把方法贴上来。
不过,如果你有钱的话,那就去买那个试剂盒吧,其中QuikChange® Site-Directed Mutagenesis Kit标准点突变试剂盒 、QuikChange® XL Site-Directed Mutagenesis Kit长模板单点突变试剂盒(>8kb)的原理我上面已经说了,只是补充了一些我认为的注意事项。如果你更有钱的话,那么你可以叫其它公司帮你做定点突变服务,大约是改一个点1000元左右。如果有需要我可以提供公司的联系方式。
下面我以一个例子为例来讲100个bp以下的碱基插入缺失或者改变实验方案。其实这种方案并不是那么好的,只不过考虑到大家一般都没有TYPEII限制性内切酶或者UDG and NTHIII(另外两种方法),所以才打算先介绍这种方法。
首先先说明一点,这种 方法在原理上存在一定成功机率,也就是说有运气成分。而定点突变则一般都是百分之百成功的,而这种100bp以下的插入缺失或者碱基改变可能要测几个克隆才能挑到一个好的克隆,大家如果要用请慎重考虑。
同样的,我只变那几十个碱基,并没有改变载体及其它地方,所以我还是依赖于DPNI酶。
举例:
Homo sapiens FzE3 是一个人类基因,其含有32个氨基酸的信号肽MRDPGAAAPLSSLGLCALVLALLGALSAGAGA,后面是成熟肽QPYHGEKGISVPDHGFCQPISIPLCTDI
AYNQTILPNLLGHTNQEDAGLEVHQFYPLVKVQCSPELRFFLCSMYAPVCTVLDQAIPPC
RSLCERARQGCEALMNKFGFQWPERLRCENFPVHGAGEICVGQNTSDGSGGPGGGPTAYP
TAPYLPDLPFTALPPGASDGRGRPAFPFSCPRQLKVPPYLGYRFLGERDCGAPCEPGRAN
GLMYFKEEERRFARLWVGVWSVLCCASTLFTVLTYLVDMRRFSYPERPIIFLSGCYFMVA
VAHVAGFLLEDRAVCVERFSDDGYRTVAQGTKKEGCTILFMVLYFFGMASSIWWVILSLT
WFLAAGMKWGHEAIEANSQYFHLAAWAVPAVKTITILAMGQVDGDLLSGVCYVGLSSVDA
LRGFVLAPLFVYLFIGTSFLLAGFVSLFRIRTIMKHDGTKTEKLEKLMVRIGVFSVLYTV
PATIVLACYFYEQAFREHWERTWLLQTCKSYAVPCPPGHFPPMSPDFTVFMIKYLMTMIV
GITTGFWIWSGKTLQSWRRFYHRLSHSSKGETAV,想在信号肽和成熟肽之间插入一个FLAG标签并在标签前面加上一个Leucine。即在信号肽和成熟肽之间插入一段序列:TTAATGGACTACAAAGACGATGACGACAAG(一共三十个bp)、
实验设计:
信号肽:
ATGCGGGACCCCGGCGCGGCCGTTCCGCTTTCGTCCCTGGGCTTCTGTGCCCTGGTGCTG
GCGCTGCTGGGCGCACTGTCCGCGGGCGCCGGGGCG
成熟肽:
CAGCCGTACCACGGAGAGAAGGGC
ATCTCCGTGCCGGACCACGGCTTCTGCCAGCCCATCTCCATCCCGCTGTGCACGGACATC
GCCTACAACCAGACCATCCTGCCCAACCTGCTGGGCCACACGAACCAAGAGGACGCGGGC
CTCGAGGTGCACCAGTTCTACCCGCTGGTGAAGGTGCAGTGTTCTCCCGAACTCCGCTTT
TTCTTATGCTCCATGTATGCGCCCGTGTGCACCGTGCTCGATCAGGCCATCCCGCCGTGT
CGTTCTCTGTGCGAGCGCGCCCGCCAGGGCTGCGAGGCGCTCATGAACAAGTTCGGCTTC
CAGTGGCCCGAGCGCCTGCGCTGCGAGAACTTCCCGGTGCACGGTGCGGGCGAGATCTGC
GTGGGCCAGAACACGTCGGACGGCTCCGGGGGCCCAGGCGGCGGGCCCACTGCCTACCCT
ACCGCGCCCTACCTGCCGGACCTGCCCTTCACCGCGCTGCCCCCGGGGGCCTCAGATGGC
AAGGGGCGTCCCGCCTTCCCCTTCTCATGCCCCCGTCAGCTCAAGGTGCCCCCGTACCTG
GGCTACCGCTTCCTGGGTGAGCGCGATTGTGGCGCCCCGTGCGAACCGGGCCGTGCCAAC
GGCCTGATGTACTTTAAGGAGGAGGAGAGGCGCTTCGCCCGCCTCTGGGTGGGCGTGTGG
TCCGTGCTGTGCTGCGCCTCGACGCTCTTTACCGTTCTCACGTACCTGGTGGACATGCGG
CGCTTCAGCTACCCAGAGCGGCCCATCATCTTCCTGTCGGGCTGCTACTTCATGGTGGCC
GTGGCGCACGTGGCCGGCTTCTTTCTAGAGGACCGCGCCGTGTGCGTGGAGCGCTTCTCG
GACGATGGCTACCGCACGGTGGCGCAGGGCACCAAGAAAGAGGGCTGCACCATCCTCTTC
ATGGTGCTCTACTTCTTCGGCATGGCCAGCTCCATCTGGTGGGTCATTCTGTCTCTCACT
TGGTTCCTGGCGGCCGGCATGAAATGGGGCCACGAAGCCATCGAGGCCAACTCGCAGTAC
TTCCACCTGGCCGCGTGGGCCGTGCCCGCCGTCAAGACCATCACTATCCTGGCCATGGGC
CAGGTAGACGGGGACCTGCTGAACGGGGTGTGCTACGTTGGCTTCTCCAGTGTGGACGCG
CTGCGGGGCTTCGTGCTGGCGCCTCTGTTCGTCTACTTCTTCATAGGCACGTCCTTCTTG
CTGGCCGGCTTCGTGTCCTTCTTCCGTATCCGCACCATCATGAAACACGACGGCACCAAG
ACCGAGAAGCTGGAGAAGCTCATGGTGCGCATCGGCGTCTTCAGCGTGCTCTACACAGTG
CCCGCCACCATCGTCCTGGCCTGCTACTTCTACGAGCAGGCCTTCCGCGAGCACTGGGAG
CGCACCTGGCTCCTGCAGACGTGCAAGAGCTATGCCGTGCCCTGCCCGCCCGGCCACTTC
CCGCCCATGAGCCCCGACTTCACCGTCTTCATGATCAAGTGCCTGATGACCATGATCGTC
GGCATCACCACTGGCTTCTGGATCTGGTCGGGCAAGACCCTGCAGTCGTGGCGCCGCTTC
TACCACAGACTTAGCCACAGCAGCAAGGGAGAGACCGCGGTATGA
插入序列
TTAATGGACTACAAAGACGATGACGACAAG
通过引物3端大于或等于18个碱基的匹配使引物与模板质粒搭配,再通过引物5端的序列来补上那三十个碱基,先用PNK酶把引物磷酸化,再用下面这两条引物把整个质粒给扩增出来,上游和下游引物就刚好把那三十个碱基给补上了,再参照引物的设计原则做一些润色,细心的朋友可以具体分析一下这两条引物。扩出来后再用DPNI酶把模板质粒去掉,再用连接酶把PCR产物的两端连接起来(虽然是平端连接 ,可是由于是同一条PCR产物的两端连接,效率会很高),转化后,测序验证,OK。
设计引物
forward primer:GGACTACAAAGACGATGACGACAAGCAGCCGTACCACGGAGAGAAG
88.5
reserve primer: ATTAACGCCCCGGCGCCCGCGGACAGT
86.9
但是由于引物的合成是由3端向5端合成,而且每合成多一个碱基的效率最多也是百分之九十九点几而不是百分之一百,所以我们拿到手的引物其实是一个混合物,比如说我们合成一条长二十个碱基的引物,实际上拿到手的是一个混合物,里面即含有二十个碱基的引物,也含 有一定比率的十九个、十八个、十七个……碱基的引物。
所以我们用这种方法做PCR时,如果连上的是足额长度的引物,那么实验也就成功了,如果连上的是少一两个碱基的引物,那么实验就失败了,不过引物当中主要的仍是足额长度的引物,所以成功机率还是蛮高的。不过送测序时就要做好准备,可能要测三五个才能拿到一个好的。
如果觉得这样不好的话,我稍后会附上用TYPEII酶或者UDG,NTHIII做的方法,它们是通过互补粘端来连接,就不存在这个问题。
下面附上详细实验过程:
第一步:设计引物;其实只要符合一般引物设计原理就行了,顺便说一下,引物一般的话,越长其质量就…………
第二步:引物PNK处理,一般合成的引物其三端是没有磷酸化的,所以我们要自己进行磷酸化,一般可以让其磷酸化过夜,不磷酸化的话最后一步连接就连不上哦。
第三步:PCR,跟基因定点突变一样,要用好的扩增酶和BUFFER,因为要把整个环高保真的圹增出来嘛;
第四步:DPNI处理,跟基因定点突变一样,要把模板去除干净。
第五步:连接,加上连接BUFFER和连接酶连接,
第六步:转化。
定点诱变(DpnI法)
1、引物设计:每条引物都要携带有所需的突变位点,引物一般长25~45bp,设计的突变位点需位于引物中部。
2、反应:使用高保真的pyrobest DNA聚合酶 ;循环次数少,一般为12个循环。
反应体系:
10x pyrobest Buffer 5 ul
dNTP Mixture(10mM) 1ul
模板DNA(5~50ng) 1ul
primer 1 (125ng) 1ul
primer 2 (125ng) 1ul
pyrobest DNA polymerase(TaKaRa)(5U/ul) 0.25ul
加无菌蒸馏水至 50ul
3、产物沉淀纯化:加1/10 体积的醋酸钠,1倍体积的异丙醇,混匀置冰上(或-20゜C冰箱)5min,离心弃上清,70~75%乙醇洗盐两次,烘干后溶于无菌水中。(此步可省略,直接用 DpnI酶切)
4、DpnI酶切:Buffer 2ul
BSA(100╳) 0.2ul
DNA x ul
DpnI 0.5ul
加无菌去离子水至 20ul
30゜C酶切 1~4 h ;65゜C 水浴15min 终止反应。
5、将酶切产物转化大肠杆菌DH5a菌株,利用抗生素筛选突变子。
6、测序验证
把目的基因上面的一个碱基换成另外一个碱基。
二、定点突变的原理
定点突变是指通过聚合酶链式反应(PCR)等方法向目的DNA片段(可以是基因组,也可以是质粒)中引入所需变化(通常是表征有利方向的变化),包括碱基的添加、删除、点突变等。定点突变能迅速、高效的提高DNA所表达的目的蛋白的性状及表征,是基因研究工作中一种非常有用的手段。
体外定点突变技术是研究蛋白质结构和功能之间的复杂关系的有力工具,也是实验室中改造/优化基因常用的手段。蛋白质的结构决定其功能,二者之间的关系是蛋白质组研究的重点之一。对某个已知基因的特定碱基进行定点改变、缺失或者插入,可以改变对应的氨基酸序列和蛋白质结构,对突变基因的表达产物进行研究有助于人类了解蛋白质结构和功能的关系,探讨蛋白质的结构/结构域。而利用定点突变技术改造基因:比如野生型的绿色荧光蛋白(wtGFP)是在紫外光激发下能够发出微弱的绿色荧光,经过对其发光结构域的特定氨基酸定点改造,现在的GFP能在可见光的波长范围被激发(吸收区红移),而且发光强度比原来强上百倍,甚至还出现了黄色荧光蛋白,蓝色荧光蛋白等等。定点突变技术的潜在应用领域很广,比如研究蛋白质相互作用位点的结构、改造酶的不同活性或者动力学特性,改造启动子或者DNA作用元件,提高蛋白的抗原性或者是稳定性、活性、研究蛋白的晶体结构,以及药物研发、基因治疗等等方面。
通过设计引物,并利用PCR将模板扩增出来,然后去掉模板,剩下来的就是我们的PCR产物,在PCR产物上就已经把这个点变过来了,然后再转化,筛选阳性克隆,再测序确定就行了。
三、引物设计原则
引物设计的一般原则不再重复。
突变引物设计的特殊原则:
(1)通常引物长度为25~45 bp,我们建议引物长度为30~35 bp。一般都是以要突变的碱基为中心,加上两边的一段序列,两边长度至少为11-12 bp。若两边引物太短了,很可能会造成突变实验失败,因为引物至少要11-12个bp才能与模板搭上,而这种突变PCR要求两边都能与引物搭上,所以两边最好各设至少12个bp,并且合成多一条反向互补的引物。
(2)如果设定的引物长度为30 bp,接下来需要计算引物的Tm值,看是否达到78℃(GC含量应大于40%)。
(3)如果Tm值低于78℃,则适当改变引物的长度以使其Tm值达到78℃(GC含量应大于40%)。
(4)设计上下游引物时确保突变点在引物的中央位置。
(5)最好使用经过纯化的引物。
Tm值计算公式:Tm=0.41×(% of GC)–675/L+81.5
注:L:引物碱基数;% of GC:引物GC含量。
四、引物设计实例
以GCG→ACG为例:
5’-CCTCCTTCAGTATGTAGGCGACTTACTTATTGCGG-3’
(1)首先设计30 bp长的上下游引物,并将A (T)设计在引物的中央位置。
Primer #1: 5’-CCTTCAGTATGTAGACGACTTACTTATTGC-3’
Primer #2: 5’-GCAATAAGTAAGTCGTCTACATACTGAAGG-3’
(2)引物GC含量为40%,L为30,将这两个数值带入Tm值计算公式,得到其Tm=75.5(Tm=0.41×40-675/30+81.5)。通过计算可以看出其Tm低于78℃,这样的引物是不合适的,所以必须调整引物长度。
(3)重新调整引物长度。
Primer #1: 5’-CCTCCTTCAGTATGTAGACGACTTACTTATTGCGG-3’
Primer #2: 5’-CCGCAATAAGTAAGTCGTCTACATACTGAAGGAGG-3’
在引物两端加5mer(斜体下划线处),这样引物的GC含量为45.7%,L值为35,将这两个数值带入Tm值计算公式,得到其Tm为80.952(Tm=0.41×47.5-675/35+81.5),这样的引物就可以用于突变实验了。
五、突变所用聚合酶及Buffer
引物和质粒都准备好后,当然就是做PCR喽,不过对于PCR的酶和buffer,不能用平时的,我们做PCR把整个质粒扩出来,延伸长度达到几个K,所以要用那些GC buffer或扩增长片段的buffer,另外,要用保真性能较好的PFU酶来扩增,防止引进新的突变。
除了使用基因定点突变试剂盒,如Stratagene和塞百盛的试剂盒,但价格昂贵。可以使用高保真的聚合酶,如博大泰克的金牌快速taq酶、Takara的PrimeSTARTM HS DNA polymerase。
六、如何去掉PCR产物
最简单的方法就是用DpnI酶,DpnI能够识别甲基化位点并将其酶切,我们用的模板一般都是双链超螺旋质粒,从大肠杆菌里提出来的质粒一般都被甲基化保护起来(除非你用的是甲基化缺陷型的菌株),而PCR产物都是没有甲基化的,所以DpnI酶能够特异性地切割模板(质粒)而不会影响PCR产物,从而去掉模板留下PCR产物,所以提质粒时那些菌株一定不能是甲基化缺陷株。
DpnI处理的时间最好长一点,最少一个小时吧,最好能有两三个小时,因为如果模板处理得不干净,哪怕只有那么一点点,模板直接在平板上长出来,就会导致实验失败。
七、如何拿到质粒
直接把通过DpnI处理的PCR产物拿去做转化就行了,然后再筛选出阳性克隆,并提出质粒,拿去测序,验证突变结果。
八、图示

九、定点突变操作步骤
[A] 诱导突变基因(PCR反应)以待突变的质粒为模板,用设计的引物及Muta-direct™酶进行PCR扩增反应,诱导目的基因突变。
1. 设计点突变引物。
[注]参考引物设计指导
2. 准备模板质粒DN A
[注]用dam+型菌株(例如DH5α菌株)作为宿主菌。在end+型菌株中常有克隆数低的现象,但是对突变效率没有影响。提取质粒DNA时我们建议您使用本公司的质粒提纯试剂盒。
3. [选项]对照反应体系(50μl反应体系)
| 10×Reaction Buffer | 5μl |
| pUC18 control plasmid(10ng/μl,total 20ng) | 2μl |
| Control primer mix(20pmol/μl) | 2μl |
| dNTP mixture(each 2.5mM) | 2μl |
| dH2O | 38μl |
| Muta-direct™ Enzyme | 1μl |
| 10×Reaction Buffer | 5μl |
| Sample plasmid(10ng/μl,total 20ng) | 2μl |
| Sample primer (F)(10pmol/μl) | 1μl |
| Sample primer (R)(10pmol/μl) | 1μl |
| dNTP mixture(each 2.5mM) | 2μl |
| dH2O | 38μl |
| Muta-direct™ Enzyme | 1μl |
[注]按如下参数设置PCR扩增条件。
| Cycles | Temperature | Reaction Time |
| 1cycle | 95℃ | 30sec |
| 15cycle | 95℃ | 30sec |
| 55℃ | 1min | |
| 72℃ | 1min per plasmid Kb |
[注] 按下列提供的PCR条件进行扩增,控制PCR循环数。注意当突变点位点超过4个时会发生突变率降低的现象。
| Mutation | Cycles |
| 1~2Nucleotide | 15cycles |
| 3Nucleotides | 18cycles |
PCR反应结束后使用Mutazyme™酶消化甲基化质粒从而选择突变质粒DNA。
1. 准备PCR反应产物
2. 加入1μl(10U/μl)Mutazyme™酶37℃温育1小时。
[注]当质粒DNA用量过多时Mutazyme™酶可能发生与样品反应不完全的现象。因此我们建议为了保证突变率请严格遵照实验步骤进行操作。如果突变率低,可以适当延长反应时间或增加Mutazyme™酶用量。
[C]转化
反应完毕后在质粒DNA上会产生缺口,当把这个质粒DNA转入E.coli中时请选择dam+型菌株,例如DH5α。
1. 将10μl样品加到50μl感受态细胞里,然后放置在冰上30分钟。
2. 接下来可以参照一般的转化步骤进行。
序列分析
通常当LB平板上出白色菌落则表明发生了突变。
为了证实这一结果,我们建议对白色单菌落进行测序分析。
先讲最简单的一个点的定点突变技术,其它较长片段的突变,删除,插入技术以后会慢慢奉上:在做实验之前,我们首先要搞清楚实验的目的和实验的原理。
实验的目的应该比较明确吧:就是要把自己的基因上面的一个碱基换成另外一个碱基。一般情况下我们会有几种可能使我们需要这样去做:
第一:我们吊出来的基因有点突变,相信这可能是大家经常会遇到的问题。基因好不容易吊出来,并装进了自己的载体,却发现有一两个碱基跟自己的预期序列或所有的公共数据库不匹配,然后暴昏。
大家实验室里面还是用Taq酶为主吧,Pfu这样的高保真酶大家应该用得不多吧,Taq酶的优点和缺点都很明显:优点就是扩增效能强,缺点就是保真性差,其错配机率是比较高的,相关数字忘了,大家可以去网上查那个数字,不过感觉如果是2000bp的基因,如果扩四五十个循环的话,很大机率会出现点突变,当然这也跟具体PCR体系里的Buffer有很大关系,详细情况这里就不讨论了。
第二:要研究基因的功能,在基因上自己选定位置更换碱基的保守序列,或者改造成不同的亚型,总之就是要人工改造碱基序列符合自己的实验需要,相信这也是那些研究基因的人经常的一种思路吧。
对于第一种情况:我们首先要分析出现碱基不匹配的位置是不是重要的位置,如果不是很重要,大可不必管它,比如说是三联密码子的最后一位,碱基的改变并没有引起相应氨基酸的改变,那么一般情况下也可以不去理它。另外,在NCBI上人类的基因的版本一直在变化,也就是说同一个基因有不同的版本,或者称不同的亚型,其碱基序列有些许的差异,只要自己克隆出来的碱基序列与其中一个相匹配,一般也就可以不做定点突变了。如果有时间没钱,那干脆重新PCR然后再克隆进自己的载体了,不过最好换个保真性好一点的酶如PFU,或者PCR循环数低一点,不过这些东西有时候也得靠运气啦。实在不行的话再来做定点突变。
对于第二种情况:这种情况下一般也就只能做定点突变了。
接下来开始聊一聊定点突变的原理吧,那个Stratagene试剂盒!上面有一个说明书,说得好像很正规,不过上面好多都是什么专利啊什么注意之类的话,看都不看,我们简明扼要地只讲实验方面,通过设计引物,并利用PCR将模板扩增出来,然后去掉模板,剩下来的就是我们的PCR产物,在PCR产物上就已经把这个点变过来了,然后再转化,筛选阳性克隆,再测序确定就行了。
大家马上就会想到几个问题了:
第一:引物怎么设计呢?
第二:模板怎么去掉呢?
第三:怎么拿到质粒呢?
对于第一个问题:怎么设计引物?
我只能讲一些原则,并举一些例子。
引物设计的原则其它贴子上都有讲,这里就不重复了:
不过这种突变引物要加上一个原则:
一般都是以要突变的碱基为中心,加上两边的一段序列,两边长度至少为11-12base pair。
若两边引物太短了,很可能会造成突变实验失败,大家应该都知道,引物至少要11-12个base pair才能与模板搭上,而这种突变PCR要求两边都能与引物搭上,所以两边最好各设至少12个base pair,并且合成多一条反向互补的引物。
这么说大家可能不是很清楚,那我就举个例子吧:
X71661.1 TATCAGGAGGAATTTGAGCACTTTCAACAAGAATTGGATAAAAAAAAAGAGGAATTCCAG 960
现有序列 TATCAGGAGGAATTTGAGCACTTTCAACAAGAATTGGATAAAAAAAAAGAGGAATTCCAG 924
*********************************************** ************
|----deletion
X71661.1 AAGGGCCACCCCGACCTCCAAGGGCAGCCTGCGGAGGAAATATTTGAGAGTGTAGGAGAT 1020
现有序列 AAGGGCCACCCCGACCTCCAAGGGCAGCCTGCGGAGGAAATATTTGAGAGTGTAGGAGAT 984(上面为目的序列,下面为现有序列:我们发现有一个A碱基的缺失,其直接结果是在表达蛋白时后面的氨基酸全部错配)
我们以它为中心设计引物:两边各至少12个碱基,左边由于含有较多的A造成引物GC%含量过低,故拉长引物使GC%含量不至过低,也使引物退火温度升高。
故合成引物CAACAAGAATTGGATAAAAAAAAAGAGGAATTCCAGAAG
并合成反向互补引物CTTCTGGAATTCCTCTTTTTTTTTATCCAATTCTTGTTG
其实也不一定要反向互补序列,只要反向引物也是两边都有大于12个碱基,同时符合引物设计的原则就行了。
引物合成公司有很多家,大家可以去寻找,不同厂家的引物在价钱质量上有一些差别,不过价钱一般都是一块多一个碱基,合成时间约为一周。
这样的结果是PCR时把整个质粒都给扩出来了,得到的PCR产物是一条链完整,另一链有缺刻的PCR产物
对于第二个问题:
怎么去掉模板呢?再简单的方法就是用DpnI酶,DpnI能够识别甲基化位点并将其酶切,我们用的模板一般都是双链超螺旋质粒,从大肠杆菌里提出来的质粒一般都被甲基化保护起来(除非你用的是甲基化缺陷型的菌株),而PCR产物都是没有甲基化的,所以DpnI酶能够特异性地切割模板(质粒)而不会影响PCR产物,从而去掉模板留下PCR产物,所以提质粒时那些菌株一定不能是甲基化缺陷株,不会那么凑巧吧,哈哈。
关于第三个问题:
直接把通过DpnI处理的PCR产物拿去做转化就行了,呵呵,然后再筛选出阳性克隆,并提出质粒,拿去测序(这个就不用我多说了吧),验证突变结果,一般都没问题的啦,我做了几十个突变了,到目前为止还没有做不出来的,呵呵,不要砸我啊。
下面讲一下具体的实验步骤以及一些实验中要注意的事情:
1、 根据现有基因设计引物;
2、 合成引物并准备好模板;
3、 PCR,
4、 DpnI处理酶切产物;
5、 转化酶切产物;
6、 筛选 阳性克隆;
7、 送测序并测全长。
最后就是庆祝啦,呵呵,没什么复杂的。
引物和质粒都准备好后,当然就是做PCR喽,不过对于PCR的酶和buffer,不能用平时的,我们做PCR把整个质粒扩出来,延伸长度达到几个K,所以要用那些GC buffer或扩增长片段的buffer,另外,要用保真性能较好的PFU酶来扩增,防止引进新的突变。
那种Quick change试剂盒分为几种不同的类型
什么QuikChange® Site-Directed Mutagenesis Kit标准点突变试剂盒 、QuikChange® XL Site-Directed Mutagenesis Kit长模板单点突变试剂盒(>8kb)从原理上是一样的,只是PCR的酶和BUFFER不一样,后面用了比较适合长片段扩增的酶和BUFFER罢了,没什么特别的东西。另外,DpnI处理的时间最好长一点,最少一个小时吧,最好能有两三个小时,因为如果模板处理得不干净,哪怕只有那么一点点,模板直接在平板上长出来,就会导致实验失败。
实验板长出来的菌有两种可能
一种是质粒DPNI没处理干净长出来的(模板),一种是PCR产物转化出来的
(突变体)
不过这两种菌长得一模一样^_^,即使提出质粒来也是一样(酶切和PCR都无法区分),除了测序,是分不出来的,
做PCR时也最好做一个负对照(不加引物),
实验管由于PCR时有引物,所以在DNPI处理前里面既含有模板又含有PCR产物,而对照管由于PCR时没放引物,所以在DPNI处理前里面只有模板。
如果两者都拿去DNPI处理
就能够证明模板已经被去除干净。
若实验顺利的话应该是:正对照长菌负对照不长菌。
如果出现正负对照都长菌,那么就是DpnI没处理好,
如果正负对照都不长菌,那么有两种可能,一种是PCR阴性,也就是说PCR出问题了,另外一个可能就是转化出问题了。要搞清楚是哪个问题,跑胶说明不了问题,那就做个转化的对照,拿试剂盒的对照实验去试感受态,马上就能知道转化有没问题。
如果正对照很多菌,负对照有几个菌,那么就是DPNI处理得不干净,这个时候就得靠运气了^_^
大家有什么问题我们可以继续讨论。
另外,如果大家既没有DpnI酶也没有好的PCR酶和BUFFER的话,那也有其它办法进行定点突变,只是麻烦一点,如果大家有需要的话,我会把方法贴上来。
对于多点突变技术及较长片段的缺失插入技术,同样的,如果大家有需要的话,我会把方法贴上来。
不过,如果你有钱的话,那就去买那个试剂盒吧,其中QuikChange® Site-Directed Mutagenesis Kit标准点突变试剂盒 、QuikChange® XL Site-Directed Mutagenesis Kit长模板单点突变试剂盒(>8kb)的原理我上面已经说了,只是补充了一些我认为的注意事项。如果你更有钱的话,那么你可以叫其它公司帮你做定点突变服务,大约是改一个点1000元左右。如果有需要我可以提供公司的联系方式。
下面我以一个例子为例来讲100个bp以下的碱基插入缺失或者改变实验方案。其实这种方案并不是那么好的,只不过考虑到大家一般都没有TYPEII限制性内切酶或者UDG and NTHIII(另外两种方法),所以才打算先介绍这种方法。
首先先说明一点,这种 方法在原理上存在一定成功机率,也就是说有运气成分。而定点突变则一般都是百分之百成功的,而这种100bp以下的插入缺失或者碱基改变可能要测几个克隆才能挑到一个好的克隆,大家如果要用请慎重考虑。
同样的,我只变那几十个碱基,并没有改变载体及其它地方,所以我还是依赖于DPNI酶。
举例:
Homo sapiens FzE3 是一个人类基因,其含有32个氨基酸的信号肽MRDPGAAAPLSSLGLCALVLALLGALSAGAGA,后面是成熟肽QPYHGEKGISVPDHGFCQPISIPLCTDI
AYNQTILPNLLGHTNQEDAGLEVHQFYPLVKVQCSPELRFFLCSMYAPVCTVLDQAIPPC
RSLCERARQGCEALMNKFGFQWPERLRCENFPVHGAGEICVGQNTSDGSGGPGGGPTAYP
TAPYLPDLPFTALPPGASDGRGRPAFPFSCPRQLKVPPYLGYRFLGERDCGAPCEPGRAN
GLMYFKEEERRFARLWVGVWSVLCCASTLFTVLTYLVDMRRFSYPERPIIFLSGCYFMVA
VAHVAGFLLEDRAVCVERFSDDGYRTVAQGTKKEGCTILFMVLYFFGMASSIWWVILSLT
WFLAAGMKWGHEAIEANSQYFHLAAWAVPAVKTITILAMGQVDGDLLSGVCYVGLSSVDA
LRGFVLAPLFVYLFIGTSFLLAGFVSLFRIRTIMKHDGTKTEKLEKLMVRIGVFSVLYTV
PATIVLACYFYEQAFREHWERTWLLQTCKSYAVPCPPGHFPPMSPDFTVFMIKYLMTMIV
GITTGFWIWSGKTLQSWRRFYHRLSHSSKGETAV,想在信号肽和成熟肽之间插入一个FLAG标签并在标签前面加上一个Leucine。即在信号肽和成熟肽之间插入一段序列:TTAATGGACTACAAAGACGATGACGACAAG(一共三十个bp)、
实验设计:
信号肽:
ATGCGGGACCCCGGCGCGGCCGTTCCGCTTTCGTCCCTGGGCTTCTGTGCCCTGGTGCTG
GCGCTGCTGGGCGCACTGTCCGCGGGCGCCGGGGCG
成熟肽:
CAGCCGTACCACGGAGAGAAGGGC
ATCTCCGTGCCGGACCACGGCTTCTGCCAGCCCATCTCCATCCCGCTGTGCACGGACATC
GCCTACAACCAGACCATCCTGCCCAACCTGCTGGGCCACACGAACCAAGAGGACGCGGGC
CTCGAGGTGCACCAGTTCTACCCGCTGGTGAAGGTGCAGTGTTCTCCCGAACTCCGCTTT
TTCTTATGCTCCATGTATGCGCCCGTGTGCACCGTGCTCGATCAGGCCATCCCGCCGTGT
CGTTCTCTGTGCGAGCGCGCCCGCCAGGGCTGCGAGGCGCTCATGAACAAGTTCGGCTTC
CAGTGGCCCGAGCGCCTGCGCTGCGAGAACTTCCCGGTGCACGGTGCGGGCGAGATCTGC
GTGGGCCAGAACACGTCGGACGGCTCCGGGGGCCCAGGCGGCGGGCCCACTGCCTACCCT
ACCGCGCCCTACCTGCCGGACCTGCCCTTCACCGCGCTGCCCCCGGGGGCCTCAGATGGC
AAGGGGCGTCCCGCCTTCCCCTTCTCATGCCCCCGTCAGCTCAAGGTGCCCCCGTACCTG
GGCTACCGCTTCCTGGGTGAGCGCGATTGTGGCGCCCCGTGCGAACCGGGCCGTGCCAAC
GGCCTGATGTACTTTAAGGAGGAGGAGAGGCGCTTCGCCCGCCTCTGGGTGGGCGTGTGG
TCCGTGCTGTGCTGCGCCTCGACGCTCTTTACCGTTCTCACGTACCTGGTGGACATGCGG
CGCTTCAGCTACCCAGAGCGGCCCATCATCTTCCTGTCGGGCTGCTACTTCATGGTGGCC
GTGGCGCACGTGGCCGGCTTCTTTCTAGAGGACCGCGCCGTGTGCGTGGAGCGCTTCTCG
GACGATGGCTACCGCACGGTGGCGCAGGGCACCAAGAAAGAGGGCTGCACCATCCTCTTC
ATGGTGCTCTACTTCTTCGGCATGGCCAGCTCCATCTGGTGGGTCATTCTGTCTCTCACT
TGGTTCCTGGCGGCCGGCATGAAATGGGGCCACGAAGCCATCGAGGCCAACTCGCAGTAC
TTCCACCTGGCCGCGTGGGCCGTGCCCGCCGTCAAGACCATCACTATCCTGGCCATGGGC
CAGGTAGACGGGGACCTGCTGAACGGGGTGTGCTACGTTGGCTTCTCCAGTGTGGACGCG
CTGCGGGGCTTCGTGCTGGCGCCTCTGTTCGTCTACTTCTTCATAGGCACGTCCTTCTTG
CTGGCCGGCTTCGTGTCCTTCTTCCGTATCCGCACCATCATGAAACACGACGGCACCAAG
ACCGAGAAGCTGGAGAAGCTCATGGTGCGCATCGGCGTCTTCAGCGTGCTCTACACAGTG
CCCGCCACCATCGTCCTGGCCTGCTACTTCTACGAGCAGGCCTTCCGCGAGCACTGGGAG
CGCACCTGGCTCCTGCAGACGTGCAAGAGCTATGCCGTGCCCTGCCCGCCCGGCCACTTC
CCGCCCATGAGCCCCGACTTCACCGTCTTCATGATCAAGTGCCTGATGACCATGATCGTC
GGCATCACCACTGGCTTCTGGATCTGGTCGGGCAAGACCCTGCAGTCGTGGCGCCGCTTC
TACCACAGACTTAGCCACAGCAGCAAGGGAGAGACCGCGGTATGA
插入序列
TTAATGGACTACAAAGACGATGACGACAAG
通过引物3端大于或等于18个碱基的匹配使引物与模板质粒搭配,再通过引物5端的序列来补上那三十个碱基,先用PNK酶把引物磷酸化,再用下面这两条引物把整个质粒给扩增出来,上游和下游引物就刚好把那三十个碱基给补上了,再参照引物的设计原则做一些润色,细心的朋友可以具体分析一下这两条引物。扩出来后再用DPNI酶把模板质粒去掉,再用连接酶把PCR产物的两端连接起来(虽然是平端连接 ,可是由于是同一条PCR产物的两端连接,效率会很高),转化后,测序验证,OK。
设计引物
forward primer:GGACTACAAAGACGATGACGACAAGCAGCCGTACCACGGAGAGAAG
88.5
reserve primer: ATTAACGCCCCGGCGCCCGCGGACAGT
86.9
但是由于引物的合成是由3端向5端合成,而且每合成多一个碱基的效率最多也是百分之九十九点几而不是百分之一百,所以我们拿到手的引物其实是一个混合物,比如说我们合成一条长二十个碱基的引物,实际上拿到手的是一个混合物,里面即含有二十个碱基的引物,也含 有一定比率的十九个、十八个、十七个……碱基的引物。
所以我们用这种方法做PCR时,如果连上的是足额长度的引物,那么实验也就成功了,如果连上的是少一两个碱基的引物,那么实验就失败了,不过引物当中主要的仍是足额长度的引物,所以成功机率还是蛮高的。不过送测序时就要做好准备,可能要测三五个才能拿到一个好的。
如果觉得这样不好的话,我稍后会附上用TYPEII酶或者UDG,NTHIII做的方法,它们是通过互补粘端来连接,就不存在这个问题。
下面附上详细实验过程:
第一步:设计引物;其实只要符合一般引物设计原理就行了,顺便说一下,引物一般的话,越长其质量就…………
第二步:引物PNK处理,一般合成的引物其三端是没有磷酸化的,所以我们要自己进行磷酸化,一般可以让其磷酸化过夜,不磷酸化的话最后一步连接就连不上哦。
第三步:PCR,跟基因定点突变一样,要用好的扩增酶和BUFFER,因为要把整个环高保真的圹增出来嘛;
第四步:DPNI处理,跟基因定点突变一样,要把模板去除干净。
第五步:连接,加上连接BUFFER和连接酶连接,
第六步:转化。
定点诱变(DpnI法)
1、引物设计:每条引物都要携带有所需的突变位点,引物一般长25~45bp,设计的突变位点需位于引物中部。
2、反应:使用高保真的pyrobest DNA聚合酶 ;循环次数少,一般为12个循环。
反应体系:
10x pyrobest Buffer 5 ul
dNTP Mixture(10mM) 1ul
模板DNA(5~50ng) 1ul
primer 1 (125ng) 1ul
primer 2 (125ng) 1ul
pyrobest DNA polymerase(TaKaRa)(5U/ul) 0.25ul
加无菌蒸馏水至 50ul
3、产物沉淀纯化:加1/10 体积的醋酸钠,1倍体积的异丙醇,混匀置冰上(或-20゜C冰箱)5min,离心弃上清,70~75%乙醇洗盐两次,烘干后溶于无菌水中。(此步可省略,直接用 DpnI酶切)
4、DpnI酶切:Buffer 2ul
BSA(100╳) 0.2ul
DNA x ul
DpnI 0.5ul
加无菌去离子水至 20ul
30゜C酶切 1~4 h ;65゜C 水浴15min 终止反应。
5、将酶切产物转化大肠杆菌DH5a菌株,利用抗生素筛选突变子。
6、测序验证
相关技术服务