组织细胞的空间关系对细胞发育、分化有重要影响。Slide-seq是一种基于原位捕获的高通量空间转录组技术,特点是无偏向性、10μm分辨率,其开发者陈飞团队以“Dissecting mammalian spermatogenesis using spatial transcriptomics”为题于2021年11月发表在Cell Reports上,利用空间转录组图谱解析精子发生的表达动力学以及糖尿病对精子生成的影响。
精子发生是精子产生的生物学过程,精子生成于睾丸的曲细精管。曲细精管总共约占睾丸总体积的 60% ~ 80%,它含有生精细胞、管周细胞和支持细胞。精子发生过程起始于精原干细胞的分化,终止于成熟的精子形成。不同的精原干细胞在曲细精管中按照特殊的细胞联系排列,形成所谓的精子发生过程。高通量单细胞RNA测序(scRNA-seq)可以捕获生殖细胞在每个发育阶段的基因表达谱的异质性,然而scRNA-seq会丢失细胞所处的空间环境,未能捕捉到生殖细胞谱系和体细胞谱系之间的空间相互作用阻碍了对精子发生的全面了解。利用Slide-seq数据,作者设计了一个计算框架,可以精确地定位单个曲细精管中的睾丸细胞类型,无偏向性地系统识别具有空间模式的基因和基因程序。结合Slide-seq和靶向原位RNA测序,作者利用空间转录组图谱解析精子发生的表达动力学,证明了小鼠和人类睾丸中精原细胞微环境的细胞组成存在显著差异。最后,对野生型和糖尿病小鼠睾丸的空间图谱进行比较,发现曲细精管的空间细胞组织破坏是糖尿病诱导雄性不育的潜在机制。
1. 绘制小鼠睾丸的空间细胞图谱
作者建立了一个工作流程以将睾丸mRNA捕获到Slide-seq阵列上(图 1A和1B)。每个阵列由具有独特空间条形码的直径10 μm珠子组成,对这些条形码进行原位测序,以将每个珠子对应到一个独特的空间位置(图 1A)。接下来,在冷冻切片期间,将冷冻睾丸薄切片(厚约10 μm)放置在空间索引珠阵列的顶部,并且让珠子捕获来自睾丸切片的mRNA。来自条形码库的后续测序数据可以唯一匹配到珠阵列上的空间坐标(图 1B)。Slide-seq阵列直径 (3 mm) 可以完全覆盖成年小鼠睾丸横截面。通过此工作流程,可以通常获得较高mRNA捕获率,平均每个珠子捕获784±25个转录本(特异分子标识符UMIs),每个阵列的平均珠数29333±1514个。
在识别细胞类型之后,作者将曲细精管的信息和生精上皮周期的不同阶段分配给每个珠子。为此,作者首先进行了拟时序分析,以沿着转录轨迹排列每个珠子。该分析概括了已知的生殖细胞发育轨迹(图 1E)。接下来,作者开发一个计算流程来对属于同一曲细精管的Slide-Seq珠进行自动分组(图1F),并基于拟时序分析来重建曲细精管图像,保留了横截面的形态细节。最后,一个生精周期可以分为几个亚阶段,每个阶段都包含不同的生殖细胞亚型关联。通过将具有已知阶段依赖性表达模式(例如Prm1)的基因表达谱投影到曲细精管的 UMAP(统一流形近似和投影)上(图 1G),将每个精管分配给四个主要阶段簇(阶段 I-III、IV-VI、VII-VIII 和 IX-XII)之一(图 1H)。
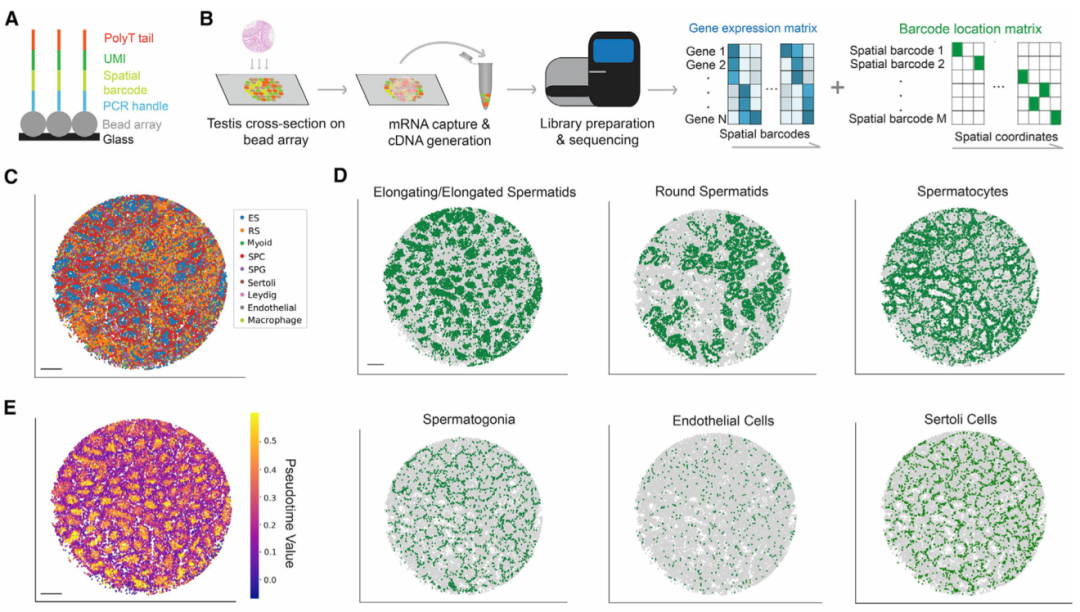
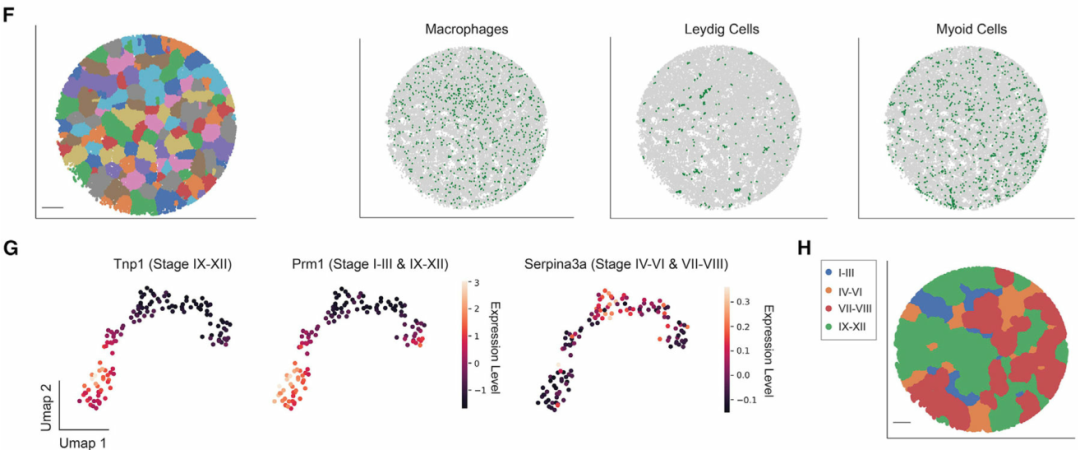
图 1 绘制小鼠睾丸空间转录组图谱,本文图片引用自Chen et al., 2021
(A) Slide-seq微珠寡核苷酸序列示意图。
(B) 睾丸样本的Slide-seq工作流程。
(C) 睾丸细胞类型的空间映射。ES,伸长中/伸长的精细胞;RS,圆形精子细胞;SPC,精母细胞;SPG,精原细胞。比例尺,300 μm。
(D) 单个睾丸细胞类型的空间映射。比例尺,300 μm。
(E) 生殖细胞发育轨迹的拟时序重建。比例尺,300 μm。
(F) 曲细精管的数字分割。比例尺,300 μm。
(G) 曲细精管在基因表达空间中的UMAP投影。细胞簇根据已知阶段特异性表达模式基因的表达量来着色。
(H) 四个阶段集群的空间映射。比例尺,300 μm。
2. 曲细精管中 SP 基因的系统鉴定
具有空间非随机分布的基因可能在不同的细胞类型或细胞亚型中发挥作用。为此,作者使用空间转录组图谱在单个曲细精管水平鉴定空间非随机分布的基因(以下称为SP基因)(图 2A)。在所有SP基因中,57.9% (589)是先前鉴定的广泛出现在多种类型细胞的基因。作者使用单分子荧光原位杂交(smFISH)证实了SP基因鉴定的准确性(图 2B)。接下来,作者对已识别的SP基因进行了基因本体(GO)富集分析。尽管精子细胞发育和分化等信号通路在所有阶段共享,但作者发现某些信号通路是阶段富集的(图 2D)。作者通过观察精细胞在精子发生过程中的基因表达谱,并根据它们所属的阶段对它们进行排序(图 2E)。作者根据各个阶段细胞的空间转录组数据进行排序,概括出精子发生过程中组蛋白到精蛋白转变的时间动态。
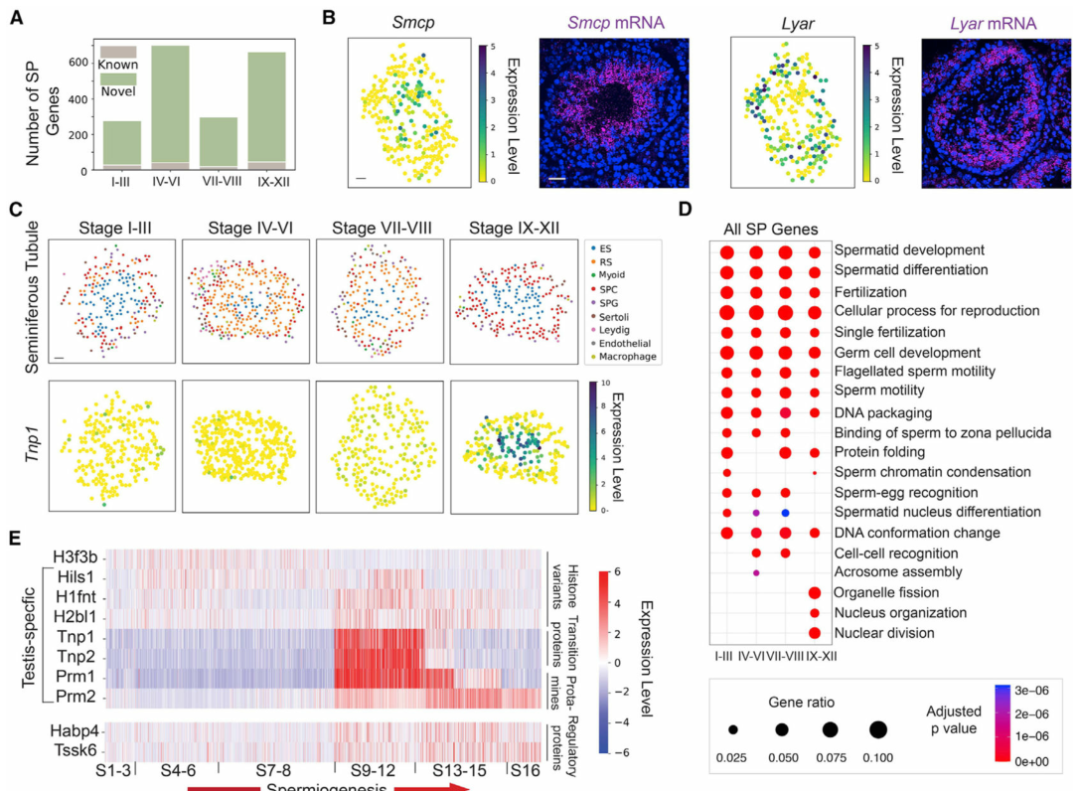
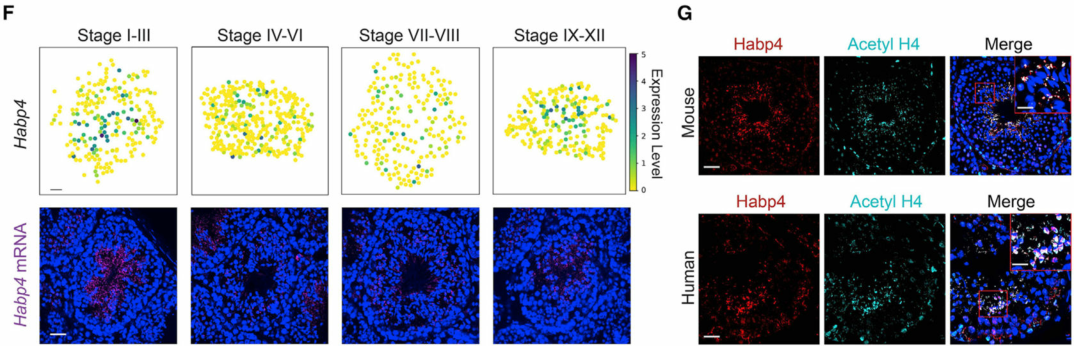
图 2 曲细精管空间非随机分布(SP)基因的系统鉴定
(A) 在生精上皮循环的每个阶段,具有先前已知空间模式的基因数量与使用空间转录组图谱新鉴定的基因数量。
(B)空间转录组图谱和单分子荧光原位杂交 (smFISH)揭示了Smcp和Lyar的空间表达模式。比例尺代表 30 μm 的数字重建曲细精管图像和 50 μm 的 smFISH 图像。
(C) 空间转录组图谱揭示了Tnp1的阶段依赖性空间表达模式。ES,伸长中/伸长的精细胞;RS,圆形精子细胞;SPC,精母细胞;SPG,精原细胞。
(D)四个阶段簇的SP基因的基因本体(GO)富集分析。
(E) 精子发生过程中核组织中富集的 SP 基因的时间表达动态。
(F) 空间转录组图谱和 smFISH 揭示了Habp4的阶段依赖性空间表达模式。
(G) Habp4 蛋白与乙酰化组蛋白 4 (Acetyl H4) 在小鼠和人类精子细胞中的共定位。比例尺代表鼠标图像的 40 μm 和插图的 10 μm。比例尺,人类图像为40 μm ,插图为15 μm。
3. 识别间质细胞和巨噬细胞的阶段特异性表达模式
作者接下来转向在不同睾丸细胞类型中具有阶段特异性表达模式的基因。阶段特异性基因表达已被证明是睾丸中生殖细胞和体细胞的基本特征。然而,间质和管周空间中体细胞的阶段特异性表达模式在很大程度上仍未明确。为此,作者使用空间转录组图谱对间质细胞和巨噬细胞进行差异基因表达分析。作者首先在I-III、IV-VI和VII-VIII 阶段簇中分别鉴定了间质细胞的阶段特异性基因(图 3A)。为了进一步验证结果,作者使用smFISH来确认H2bl1在 IV-VI 期相关间质细胞中的阶段特异性(图 3B)。有趣的是,作者注意到表达H2bl1(也称为1700024P04Rik)的间质细胞优先定位在管周间隙附近(图 3B)。表达H2bl1的间质细胞的稀疏性和空间定位类似于干间质细胞,作者发现H2bl1可能参与调节间质干细胞功能。与对间质细胞的分析类似,对巨噬细胞群进行差异基因表达分析(图 3C)。然后作者使用smFISH来确认Efcab6的阶段特异性表达模式(图 3D)。作者通过鉴定阶段依赖性基因表达程序,证明了生精上皮细胞的循环如何影响间质细胞和巨噬细胞的分子组成,还根据空间定位成功区分了睾丸巨噬细胞的两个谱系。
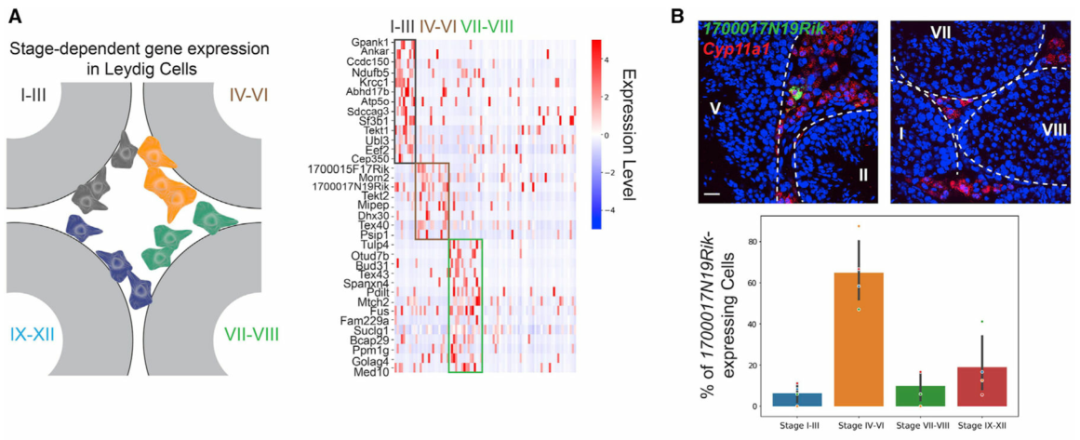
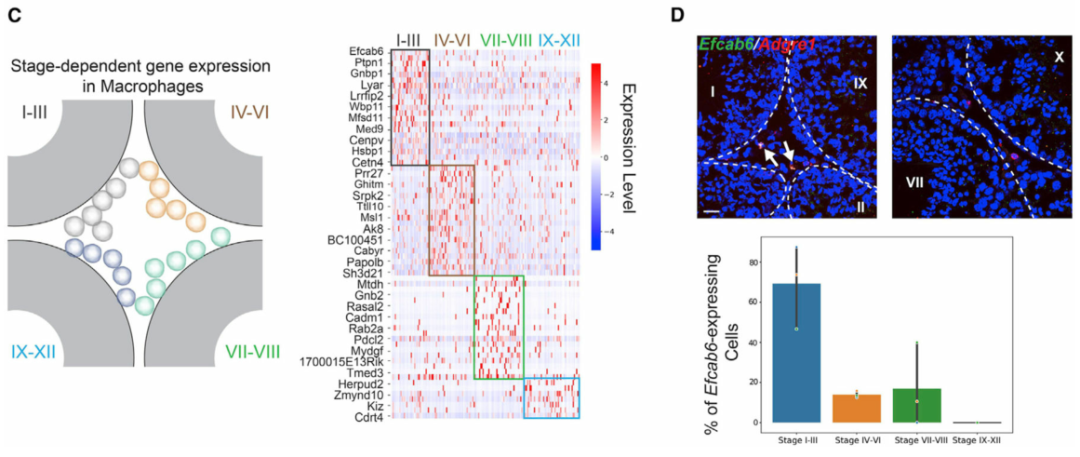
图 3 间质细胞和巨噬细胞的阶段依赖性基因表达模式
(A) 左图:间质细胞空间定位示意图。右图:在间质细胞中表现出阶段依赖性表达模式的基因。
(B) 上图:表达1700017N19Rik的间质细胞(由Cyp11a1标记)定位在 IV-VI 期曲细精管附近,但在其他阶段不靠近小管。基底膜由白色虚线勾勒。比例尺,20 μm。下图:总结不同阶段组曲细精管附近表达1700017N19Rik的间质细胞百分比的条形图。
(C) 左:巨噬细胞空间定位示意图。右图:巨噬细胞中表现出阶段依赖性表达模式的基因。
(D)上图:表达Efcab6的巨噬细胞(由Adgre1标记)定位在 I-III 期曲细精管附近,但在其他阶段不靠近小管。基底膜由白色虚线勾勒。比例尺,20 μm。下图:条形图总结了不同阶段组曲细精管附近表达Efcab6的巨噬细胞的百分比。
4. 原位RNA测序分析精原细胞微环境的空间
精原细胞 (SPG) 包含干细胞群,以往的研究表明,SPG的自我更新和分化受其周围微环境的影响。为了系统地分析精原细胞微环境,作者首先在空间上绘制了小鼠(图 4A)和人类(图 4E)睾丸中未分化和分化 SPG 的种群。令人惊讶的是,与小鼠相比,作者发现人类未分化和分化SPGs周围微环境的空间细胞组成存在显著差异(图 4F)。对于睾丸间质细胞、内皮细胞和巨噬细胞等体细胞类型尤其如此,但对于支持细胞和肌样细胞则不然,这表明在人类睾丸中这些体细胞类型之间在塑造精原细胞微环境方面可能存在不同的作用。最后,作者使用多重smFISH证实了这一发现,显示了人类未分化和分化SPG周围微环境中内皮细胞的差异富集(图 4G)。作者的数据揭示了小鼠和人类精原细胞微环境空间结构的显著差异,表明两种物种在精子发生的早期阶段存在不同的调控机制。
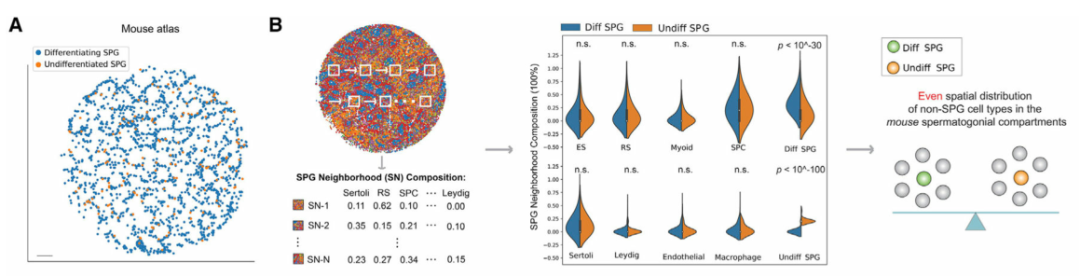
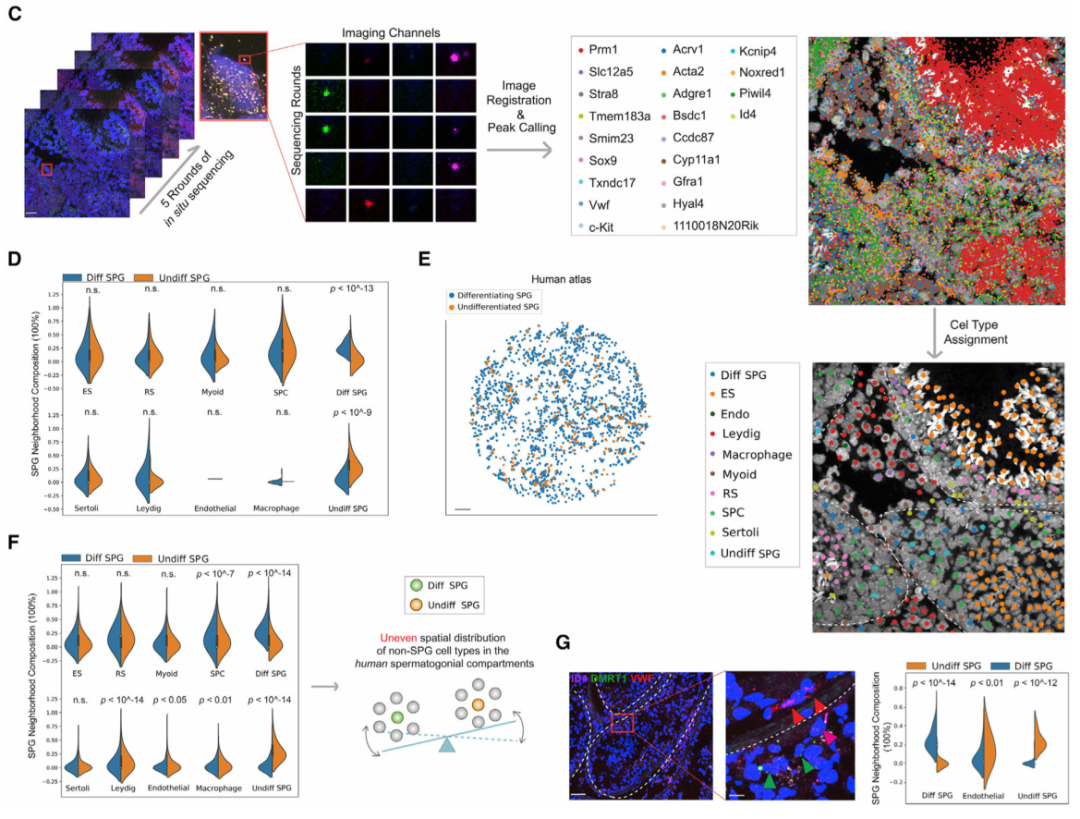
图 4 小鼠与人类睾丸中的差异干细胞微环境
(A)使用 Slide-seq 数据对小鼠未分化和分化精原细胞进行空间映射。比例尺,300 μm。
(B) K 最近邻 (KNN) 方法,使用 Slide-seq 数据计算每个小鼠精原细胞周围微环境中的细胞类型组成。此处显示了未分化精原细胞与具有 K = 5 个邻居的分化精原细胞周围微环境的细胞类型组成的比较。显示 K = 10 和 15 的图显示在图 S5B中。图 S5C中显示了两个额外的生物学重复。ns,不显著;ES,伸长中/伸长的精细胞;RS,圆形精子细胞;SPC,精母细胞;Diff SPG,区分精原细胞;Undiff SPG,未分化的精原细胞。
(C)针对 22 个基因的小鼠睾丸样本的原位测序。ES,伸长中/伸长的精细胞;RS,圆形精子细胞;Diff SPG,区分精原细胞;Undiff SPG,未分化的精原细胞;内皮细胞。白色虚线勾勒出图像中的曲细精管。比例尺代表 20 μm(插图为 2 μm)。
(D)使用 (C) 中生成的数据计算未分化小鼠与分化精原细胞周围微环境的细胞类型组成的 KNN。使用了 K = 5 个邻居。显示 K = 10 和 15 的图显示在图 S5E中。ns,不显著;ES,伸长中/伸长的精细胞;RS,圆形精子细胞;SPC,精母细胞;Diff SPG,区分精原细胞;Undiff SPG,未分化的精原细胞。
(E)使用 Slide-seq 数据对人类未分化和分化精原细胞进行空间映射。比例尺,300 μm。
(F)人类未分化与分化精原细胞周围微环境的细胞类型组成的比较。使用了 K = 10 个邻居。图 S5G显示了显示 K = 15 以及不同复制的图。ns,不显著;ES,伸长/伸长的精子细胞;RS,圆形精子细胞;SPC,精母细胞;Diff SPG,区分精原细胞;Undiff SPG,未分化的精原细胞。
(G)使用多路复用 smFISH 数据比较人类未分化与分化精原细胞周围微环境的内皮细胞组成。Diff SPG,区分精原细胞;Undiff SPG,未分化的精原细胞。红色箭头,内皮细胞;洋红色箭头,未分化的精原细胞;绿色箭头,区分精原细胞。白色虚线勾勒出图像中的曲细精管。比例尺代表 70 μm(插图为 10 μm)。
5. 糖尿病通过破坏曲细精管的空间结构诱发睾丸损伤
最后,作者设想Slide-seq工作流程和计算流程也可以应用于分析精子发生的病理变化。为此,作者从三只瘦素缺陷型糖尿病小鼠 ( ob/ob ) 和三只对照的野生型 (WT) 小鼠中生成了Slide-seq数据。糖尿病(DM)的一个重要并发症是男性生殖系统的紊乱,然而其潜在机制尚未得到充分认识。作者在空间上绘制已识别的表达差异基因的表达。通过放大单个曲细精管,作者发现Smcp的空间表达模式在ob/ob曲细精管中被破坏(图 5B)。由于已经证明Smcp的空间表达模式不依赖于阶段变化,因此这种破坏不太可能是由于生精上皮周期阶段的差异。对ob/ob小管的仔细检查表明,基因表达模式的变化部分是由于空间细胞组织的改变。在 WT 生精管中,ES(伸长中/伸长的精细胞)在中心聚集在一起,显示出较高的平均纯度评分(图 5C,左图)。相比之下,这种空间组织在ob/ob曲细精管中被破坏,其中ES更有可能与其他类型细胞进行空间接触(图 5C,右图)。作者观察到ES和所有其他类型细胞之间的空间排列发生了显著变化(图 5F)。这表明在糖尿病条件下,精子细胞的空间排列最容易受到破坏性影响。总之,作者的分析表明,曲细精管空间结构的破坏是糖尿病引起的睾丸损伤的潜在机制。
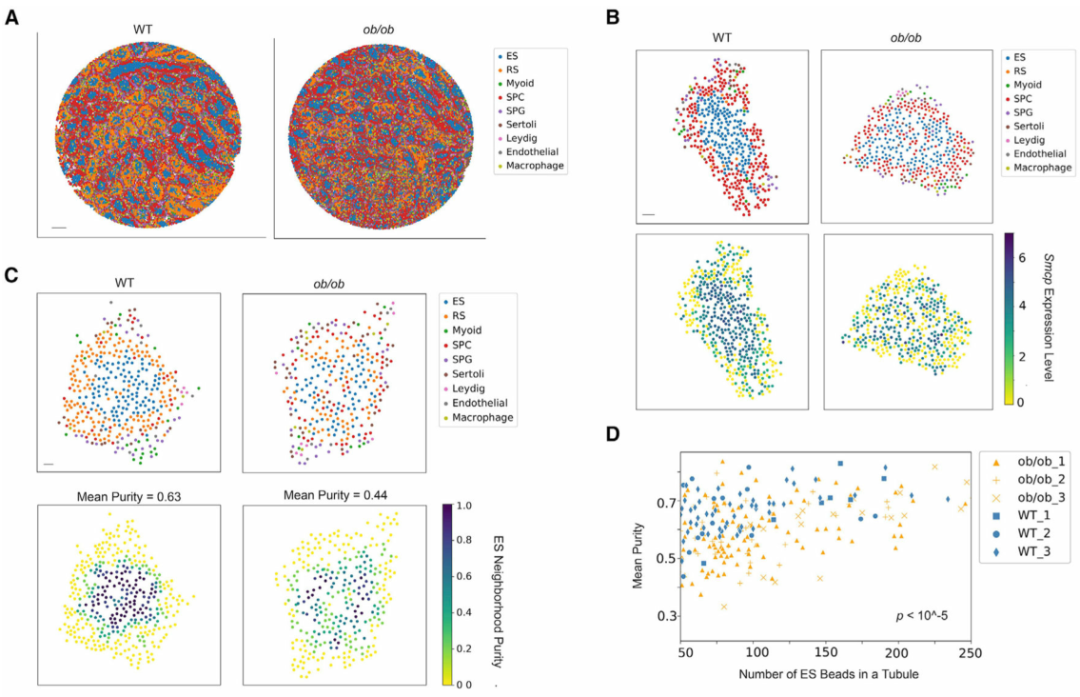
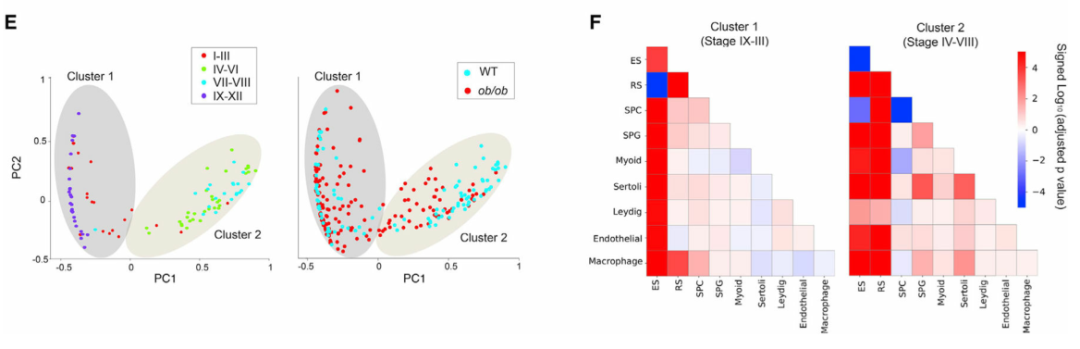
图 5 糖尿病破坏曲细精管中睾丸细胞类型的空间排列
(A)野生型 (WT) 和ob/ob样本睾丸细胞类型的空间映射。比例尺,500 μm。
(B) Smcp的空间表达模式在有代表性的ob/ob曲细精管中被破坏。ES,伸长中/伸长的精细胞;RS,圆形精子细胞;SPC,精母细胞;SPG,精原细胞。比例尺,30 μm。
(C)代表 WT 和ob/ob曲细精管中每个Slide-seq珠子的 ES 纯度分数,分别。还显示了每个小管中具有非零值的珠子的平均纯度分数。比例尺,30 μm。
(D)来自 WT 和 ob/ob 样本的每个曲细精管至少有 50 个 ES珠子的平均纯度评分。使用 Mann-Whitney U检验对两种条件之间的纯度评分进行比较。
(E)左:WT曲细精管中成对空间接触频率的主要成分揭示的两个 WT 曲细精管结构簇。标记每个精管的阶段信息。右图:ob/ob曲细精管投影到左图中所示的两个簇上。
(F) Mann-Whitney U检验对两个簇下 WT 和ob/ob曲细精管之间的成对空间细胞接触频率。显示了细胞类型之间空间接触频率显著增加(正)和减少(负)的有符号 p 值。
在小鼠和人类睾丸中,睾丸细胞在曲细精管内和周围以特定空间组织支持精子发生。在这项研究中,作者分别在空间上绘制了小鼠和人类曲细精管中未分化和分化的精原细胞(SPG)细胞图谱,微环境分析确定了小鼠和人类睾丸之间未分化和分化精原细胞周围细胞组成模式的主要差异。关于这些差异如何与这两个物种中精原干细胞活性的差异调节相关的研究可能会为哺乳动物精子发生的调节机制提供见解。除了分析正常睾丸样本外,作者还系统地比较了WT和糖尿病曲细精管之间的空间细胞结构。通过计算不同细胞类型之间的空间混合程度和成对细胞接触频率,作者观察到糖尿病曲细精管的空间细胞组织存在显著破坏,实验证明细胞和SPG之间的空间接触频率发生了显著变化。证明了曲细精管水平的空间细胞组织的破坏可能是糖尿病损害雄性生育能力的一种机制。
参考文献
Chen H, Murray E, Sinha A, et al. Dissecting mammalian spermatogenesis using spatial transcriptomics. Cell Rep. 2021;37(5):109915. doi:10.1016/j.celrep.2021.109915