技术专题
知无不“研”|一文读懂染色质免疫共沉淀技术(ChIP)
文章来源:genecreate
作者:genecreate
发布时间:2020-06-08 17:01
基于体内分析而发展的染色质免疫沉淀分析(Chromatin immunoprecipitation assay kit,ChIP)技术可以真实、完整地反映结合在DNA序列上的调控蛋白。由于ChIP采用甲醛固定活细胞或者组织的方法,因此能比较真实的反映细胞内TF与Promoter的结合情况,还可以用来研究组蛋白的各种共价修饰与基因表达的关系。近年来,这种技术得到不断的发展和完善。采用结合微阵列技术在染色体基因表达调控区域检查染色体活性,是深入分析癌症、心血管病以及中央神经系统紊乱等疾病主要通路的一种非常有效的工具。
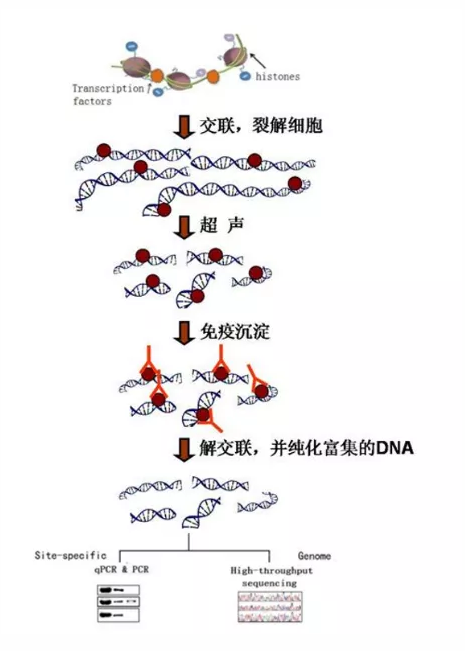
染色质免疫沉淀分析(ChIP)的基本原理是在活细胞状态下,当用甲醛处理时,相互靠近的蛋白与蛋白、蛋白与核酸(DNA或RNA)之间会产生共价键。细胞内,当TF与Promoter相互结合时,它们必然靠的比较近或者契合在一起,这个时候用甲醛处理,能使它们之间产生共价键。固定的蛋白质-DNA复合物通过超声或酶处理将其随机切断为一定长度范围内的染色质小片段,然后通过抗原抗体的特异性识别反应沉淀此复合体,特异性地富集目的蛋白结合的DNA片段,通过对目的片断的纯化与检测,从而获得蛋白质与DNA相互作用的信息。通过qPCR或二代测序,筛选与目的蛋白互作的未知DNA信息。
今天小编将珍藏多年的ChIP实验心得拿出来与大家一同探讨。
应用领域
1、判断DNA链的某一特定位置会出现何种组蛋白修饰
2、检测RNA polymerase II及其它反式因子在基因组上结合位点的精确定位
3、研究组蛋白共价修饰与基因表达的关系
4、转录因子研究
技术流程
案例展示
案例一
题目:Hypoxia induces H19 expression through direct and indirect Hif-1α activity, promoting oncogenic effects in glioblastoma
期刊:scientific reports
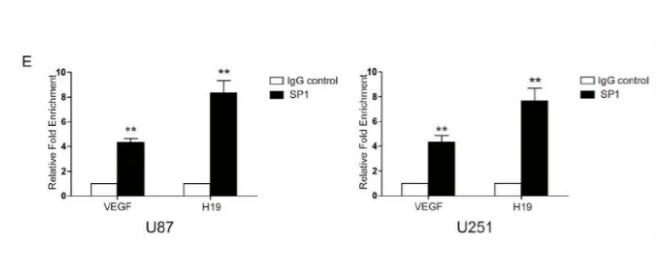
主要内容:作者分别使用了两种细胞U87和U251,验证SP1对于H19的转录调控作用。用SP1抗体进行ChIP实验,对H19的启动子区域上游100bp的高GC含量,设计引物。富集到的DNA进行qPCR检测,结果显示SP1能够调控H19启动子的转录。
案例二
题目:Chromatin Profiling by Directly Sequencing Small Quantities of Immunoprecipitated DNA.
期刊:Nat Methods
主要内容:收集培养的5×106K562细胞,使用组蛋白甲基化修饰抗体H3K27me3、H3K4me3、H3K36me3(ChIP-grade)进行IP富集提取的经过片段化的染色质复合物,构建测序文库后进行二代测序,获得peaks在染色质上的分布情况。
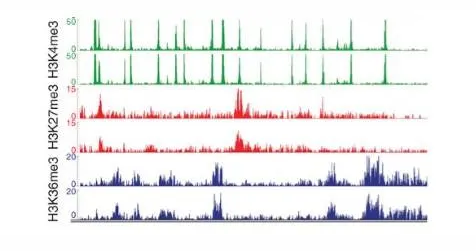
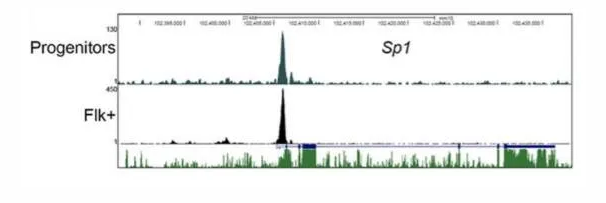
案例三
题目: A crucial role for the ubiquitously expressed transcription factor Sp1 at early stages of hematopoietic specification.
期刊:Development
主要内容:分离培养小鼠胚胎干细胞并诱导分化,分别收集组细胞及Flk+细胞,用转录因子Sp1抗体进行ChIP实验后二代测序,获得Sp1结合位点在两种细胞中的分布区域。
ChIP经验分享
ChIP实验操作性很强,金开瑞根据多年的实践,总结了一些ChIP实验中您可能会遇到的棘手的问题,下面我们就一起来康康吧!
细胞固定
1、甲醛终浓度为1%较适宜
2、固定时间一般为5-60min较好,具体时间根据实验而定
染色质断裂
1、 超声时断时续,保证低温
2、 注意探头位置,防止产生泡沫
3、 研究组蛋白需使用酶处理
染色质免疫沉淀
1、 最好有Input对照。Input对照不仅可以验证染色质断裂的效果,还可以根据Input中的靶序列的含量以及染色质沉淀中的靶序列的含量,按照取样比例换算出ChIP的效率,所以Input对照是ChIP实验必不可少的步骤。
2、 抗体的选择是关键。抗体的选择要注意三点:一是应该选择能够做ChIP实验的抗体。尽量选择ChIP级别商业化的抗体。如没有,一般可以重组到带有标签的载体上,再转染相应的宿主(目前市面上商业化的chip抗体只有300多种,因此正好找到对应抗体较困难,可以选择标签抗体)。二是抗体的结合位点。选择单抗的话,尽量远离抗原和染色质相结合的位点,这样可以最大限度保证抗体和抗原的结合,而多抗可识别多个表位,可基本避免该风险。因此单多抗各有优缺点,应重点关注该抗体是否经过ChIP验证。三是抗体的浓度。抗体浓度的也很重要,浓度太低,不能与靶蛋白完全结合。浓度太高会导致非特异性条带增加背景。
3、 阴性对照可以使用血清或lgG,阳性对照一般使用RNA Polymerase II或者组蛋白。
阴性对照:用实验抗体同型的IgG作为抗体,理论上不会ChIP下来任何DNA片段,因此作为阴性对照,但是由于非特异结合,或者实验过程中,没发生结合的DNA清除不完全,可能也会出现条带。如果阴性对照样品中的产物量等于特异靶标样品中产物量,说明特异靶标抗体未发挥作用或者染色质断裂不充分。
阳性对照:为了保证阳性对照有效,一般选择阳性对照的引物是根据管家基因设计的。一般用RNA Polymerase II或者Histone H3(组蛋白)抗体,因为RNA Polymerase II是通用转录因子,在所有细胞中都能结合基因的核心启动子区。因此,理论上ChIP后PCR都会有条带。
4、 需设计已经确定的、不与特异抗原相结合的DNA片段的引物,用来排除抗原和染色质的非特异结合。
交联反应的逆转
1、 RNA酶、蛋白酶需65℃保温6小时
2、 不加蛋白酶可以提取、分析结合蛋白
DNA的纯化
最好用试剂盒,便于后面的PCR检测
DNA的鉴定
可以进行二代测序或者qPCR检测结果
因为ChIP实验涉及的步骤多,结果的重复性较低,所以对ChIP实验过程的每一步都应设计相应的对照,而且对结果的分析也需要有一定的经验。如果ChIP实验方面有什么问题,欢迎与我们交流哦!
实验Q&A
Q:ChIP-Seq和ChIP-qPCR有何异同?
A:染色质免疫共沉淀(ChIP)所获得的DNA产物,在ChIP-Seq中通过高通量测序的方法,在全基因组范围内寻找目的蛋白(转录因子、修饰组蛋白)的DNA结合位点片段信息;ChIP-qPCR需要预设待测的目的序列,针对目的序列设计引物,以验证该序列是否同实验蛋白结合互作。
Q:染色质片段大小在哪个范围比较合适?
A:对于ChIP-seq,片段在200-500 bp左右是最合适范围;对于ChIP-qPCR,片段在200-800 bp左右适宜。
Q:植物样本处理和动物组织/细胞有何区别?
A:植物组织由于细胞壁、气腔等结构的存在,会给交联缓冲液的作用带来困难,因此相对于动物组织/细胞来说,往往需要在抽真空条件下进行交联,而该步奏是一个需要经验及优化的过程。
Q:ChIP-Seq中的测序DNA样本需要多少产量?
A:通常是≥10ng。
Q:ChIP风险如果判断
A:ChIP实验以标签来判断实验风险,重组标签的转录因子>内源转录因子>组蛋白;当以重组蛋白作为靶蛋白时,重组蛋白同内源蛋白可能存在结合活性、结合位点差异;以标签抗体进行ChIP时、染色质结合位点本身会被内源蛋白竞争,这些都会影响到ChIP过程的特异性捕获效率。


了解更多实验Q&A请点击公众号菜单栏
猜你喜欢
讲座回放

讲座预告

小程序会员专享
ChIP-qPCR及ChIP-seq签单一律6.5折
活动时间:2020年6月1号-7月1号
详情请咨询当地销售